Investigación de la gripe española - Spanish flu research
| Virus de la influenza A subtipo H1N1 | |
|---|---|
|
|
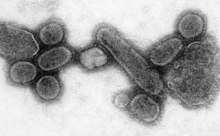
| Una micrografía electrónica del virus que causó la gripe de 1918. | |
|
Clasificación de virus |
|
| (no clasificado): | Virus |
| Reino : | Riboviria |
| Reino: | Orthornavirae |
| Filo: | Negarnaviricota |
| Clase: | Instoviricetos |
| Pedido: | Articulavirales |
| Familia: | Ortomixoviridae |
| Género: | Virus alfainfluenza |
| Especies: | |
| Serotipo: |
Virus de la influenza A subtipo H1N1
|
| Cepas muestreadas | |
|
|
La investigación sobre la influenza española se refiere a estudios sobre las causas y características de la " influenza española " , una variedad de influenza que en 1918 fue responsable de la peor pandemia de influenza en la historia moderna. Muchas teorías sobre los orígenes y avances de la gripe española persistieron en la literatura, pero no fue hasta 2005, cuando se recuperaron varias muestras de tejido pulmonar de soldados estadounidenses de la Primera Guerra Mundial y de una mujer inupiat enterrada en permafrost en una fosa común en Brevig Mission, Alaska , que hizo posible una importante investigación genética.
Origen del virus
Hay dos teorías predominantes que se postulan habitualmente. Una de las teorías de Alfred W. Crosby es que la cepa del virus se originó en Fort Riley , Kansas , por dos genéticas mecanismos - la deriva genética y el cambio antigénico - en los virus en aves de corral y cerdos, que la fortaleza criados para el consumo local. Aunque los datos iniciales de una reconstrucción reciente del virus sugirieron que saltó directamente de las aves a los humanos , sin viajar a través de los cerdos, desde entonces esto se ha puesto en duda. Un investigador publicado en 2004 argumentó que la enfermedad se encontró en el condado de Haskell, Kansas , ya en enero de 1918. Un virus similar e incluso más mortal se había visto antes en los campamentos británicos en Francia y en Aldershot.
A principios de trabajo de investigación publicado en 2000 por un equipo dirigido por el virólogo británico, John Oxford del hospital de San Bartolomé y el Royal London Hospital , sugirieron que un director de campo provisional de tropas británicas en Étaples , Francia, estuvo en el centro de la pandemia de gripe de 1918 o por lo al menos un virus precursor significativo. Hubo una misteriosa infección respiratoria en la base militar durante el invierno de 1915-1916.
Descubrimiento de genomas virales

En 1995, Jeffery Taubenberger, del Instituto de Patología de las Fuerzas Armadas de EE. UU. (AFIP), se preguntó si sería posible recuperar el virus de la pandemia de gripe de 1918 a partir del tejido seco y fijo de las víctimas. Él y sus colegas analizaron 10 portaobjetos de muestra de tejido y 2 dieron positivo. Taubenberger, Ann H. Reid y Thomas G. Fanning pudieron amplificar segmentos cortos del ácido nucleico viral mediante la reacción en cadena de la polimerasa (PCR) . Los resultados se publicaron en la revista Science en marzo de 1997.
El 20 de agosto de 1997, Johan Hultin recuperó muestras de la influenza de 1918 del cadáver congelado de una mujer nativa de Alaska enterrada durante casi ocho décadas en permafrost cerca de Brevig Mission, Alaska . Llevó las muestras a un equipo en Rockville, Maryland dirigido por Jeffery Taubenberger del Instituto de Patología de las Fuerzas Armadas de los Estados Unidos (AFIP). Brevig Mission perdió aproximadamente el 85% de su población a causa de la gripe de 1918 en noviembre de 1918. Una de las cuatro muestras recuperadas contenía material genético viable del virus. Esta muestra brindó a los científicos una oportunidad de primera mano para estudiar el virus, que se inactivó con tiocianato de guanidinio antes del transporte. Esta muestra y otras encontradas en los archivos de la AFIP permitieron a los investigadores analizar por completo las estructuras genéticas críticas del virus de 1918.
- "Ahora hemos identificado tres casos: el caso Brevig Mission y dos casos de archivo que representan las únicas fuentes conocidas de material genético del virus de la influenza de 1918", dijo Taubenberger, jefe de la división de patología molecular de la AFIP e investigador principal del proyecto.
Las muestras de autopsia archivadas habían sido tomadas de los soldados soldados de la Primera Guerra Mundial Roscoe Vaughan y James Downs.
La edición del 6 de febrero de 2004 de la revista Science informó que dos equipos de investigación, uno dirigido por Sir John Skehel, director del Instituto Nacional de Investigación Médica en Londres , otro por el profesor Ian Wilson del Instituto de Investigación Scripps en San Diego , habían logrado sintetizar la proteína hemaglutinina responsable del brote de gripe de 1918. Lo hicieron juntando ADN de una muestra de pulmón de una mujer inuit enterrada en la tundra de Alaska y varias muestras conservadas de soldados estadounidenses de la Primera Guerra Mundial. Los equipos analizaron la estructura del gen y descubrieron cómo las sutiles alteraciones en la forma de una molécula de proteína le habían permitido pasar de las aves a los humanos con efectos tan devastadores.
El 5 de octubre de 2005, Tumpey y otros investigadores de los Centros para el Control y la Prevención de Enfermedades (CDC) en Atlanta , Georgia, y la Escuela de Medicina Mount Sinai en Nueva York , anunciaron que la secuencia genética (~ 13 kpb) de la gripe de 1918 La cepa, un subtipo de la cepa aviar H1N1 , se había reconstruido utilizando muestras de tejido históricas y una pequeña parte del ARN de una cepa moderna.
Características del virus
Los virus de la influenza tienen una tasa de mutación relativamente alta que es característica de los virus de ARN . El virus H5N1 ha mutado en una variedad de tipos con diferentes perfiles patogénicos; algunos patógenos para una especie pero no para otras, algunos patógenos para múltiples especies. La capacidad de varias cepas de influenza para mostrar selectividad de especie se debe en gran parte a la variación en los genes de hemaglutinina. Las mutaciones genéticas en el gen de la hemaglutinina que causan sustituciones de un solo aminoácido pueden alterar significativamente la capacidad de las proteínas de hemaglutinina vírica para unirse a receptores en la superficie de las células huésped. Tales mutaciones en los virus aviar H5N1 pueden cambiar las cepas de virus de ser ineficientes para infectar células humanas a ser tan eficientes en causar infecciones humanas como los tipos más comunes de virus de influenza humana.
En julio de 2004, investigadores dirigidos por H. Deng del Instituto de Investigación Veterinaria de Harbin , Harbin , China, y Robert Webster del Hospital de Investigación Infantil St. Jude , Memphis, Tennessee , informaron los resultados de experimentos en los que los ratones habían estado expuestos a 21 aislamientos. de cepas confirmadas de H5N1 obtenidas de patos en China entre 1999 y 2002. Encontraron "un patrón temporal claro de patogenicidad progresivamente creciente". Los resultados informados por Webster en julio de 2005 revelan una mayor progresión hacia la patogenicidad en ratones y una mayor propagación del virus por parte de los patos.
En diciembre de 2008, una investigación de Yoshihiro Kawaoka de la Universidad de Wisconsin mostró que la presencia de tres genes específicos (denominados PA, PB1 y PB2) y una nucleoproteína derivada de las muestras de gripe H1N1 1918 fue suficiente para desencadenar síntomas similares en pruebas con animales.
Investigación de patogénesis viral
Investigaciones recientes de Taubenberger et al. ha sugerido que el virus de 1918, como el H5N1, podría haber surgido directamente de un virus de influenza aviar. Sin embargo, investigadores de la Universidad de Virginia y la Universidad Nacional de Australia han sugerido que puede haber una interpretación alternativa de los datos utilizados en Taubenberger et al. papel. Taubenberger y col. respondió a estas cartas y defendió su interpretación original.
Otra investigación de Tumpey y sus colegas que reconstruyeron el virus H1N1 de 1918 llegó a la conclusión de que eran los genes de la polimerasa y los genes HA y NA los que causaban la virulencia extrema de este virus. El 18 de enero de 2007, Kobasa et al. informó que los monos infectados ( Macaca fascicularis ) exhibieron síntomas clásicos de la pandemia de 1918 y murieron a causa de una tormenta de citocinas .
Las secuencias de las proteínas de la polimerasa (PA, PB1 y PB2) del virus de 1918 y los subsiguientes virus humanos difieren en solo 10 aminoácidos de los virus de la influenza aviar. Ya se han identificado virus con 7 de los 10 aminoácidos en las ubicaciones de la influenza humana en el virus H5N1 que circula actualmente . Esto ha llevado a algunos investigadores a sugerir que otras mutaciones pueden surgir y hacer que el virus H5N1 sea capaz de transmitirse de persona a persona.
Otro factor importante es el cambio de la proteína HA a una preferencia de unión por el ácido alfa-2,6 siálico (la forma principal que se encuentra en el tracto respiratorio humano). En el virus aviar, la proteína HA se une preferentemente al ácido alfa-2,3 siálico, que es la forma principal en el tracto entérico aviar. Se ha demostrado que solo un cambio de un solo aminoácido puede provocar el cambio de esta preferencia de unión. En total, es posible que solo sea necesario producir un puñado de mutaciones para que la gripe aviar H5N1 se convierta en un virus pandémico como el de 1918. Sin embargo, es importante tener en cuenta que la probabilidad de mutación no indica la probabilidad de la evolución de un virus así. cepa, ya que algunas de las mutaciones necesarias pueden verse limitadas por la selección estabilizadora .
Plasma sanguíneo como tratamiento eficaz
En caso de otra pandemia, investigadores militares estadounidenses han propuesto reutilizar un tratamiento de la mortal pandemia de 1918 para mitigar los efectos de la gripe: algunos médicos militares inyectaron a pacientes gravemente afectados con sangre o plasma sanguíneo de personas que se habían recuperado de la gripe. gripe. Los datos recopilados durante ese tiempo indican que el tratamiento con inyección de sangre redujo las tasas de mortalidad hasta en un 50 por ciento.
Los investigadores de la Marina han lanzado una prueba para ver si el tratamiento de 1918 funcionará contra la mortal gripe aviar asiática. Los resultados hasta ahora no han sido concluyentes. El plasma humano H5N1 puede ser un tratamiento eficaz, oportuno y ampliamente disponible para la próxima pandemia de gripe. Un nuevo estudio internacional que utilice métodos modernos de recopilación de datos sería un proceso lento y difícil. Citando la espera de meses para una vacuna para la próxima pandemia, muchos expertos en gripe opinan que el método de 1918 es algo a considerar.
En la pandemia mundial de gripe de 1918 , "los médicos probaron todo lo que sabían, todo lo que habían oído hablar, desde el antiguo arte de sangrar a los pacientes hasta la administración de oxígeno y el desarrollo de nuevas vacunas y sueros (principalmente contra lo que ahora llamamos Hemophilus influenzae , un nombre derivado del hecho de que originalmente se consideró el agente etiológico (y varios tipos de neumococos). Solo una medida terapéutica mostró algún indicio de éxito: transfundir sangre de pacientes recuperados a nuevas víctimas ".
Ver también
- Investigación sobre la influenza
- Mark Sykes - exhumación de una víctima de la gripe británica en el Reino Unido
- Yoshihiro Kawaoka : diseñó y recreó un virus para estudiar cómo funciona y cómo muta naturalmente la gripe.

