ARN nucleolar pequeño U3 - Small nucleolar RNA U3
| U3 | |
|---|---|
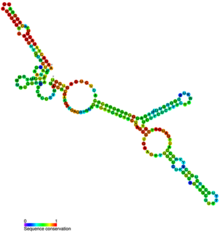 Conservación de la secuencia y estructura secundaria del ARN U3 del metazoo
| |
| Identificadores | |
| Símbolo | U3 |
| Alt. Simbolos | RNU3P2, |
| Rfam | RF00012 |
| Gen NCBI | 26844 |
| HGNC | 10176 |
| OMIM | 180710 |
| Otros datos | |
| Tipo de ARN | snoRNA |
| Dominio (s) | Eucariota ; |
| Estructuras PDB | PDBe |
El ARNsno U3 es un ARN no codificante que se encuentra predominantemente en el nucleolo . U3 tiene motivos de caja C / D que técnicamente lo hacen miembro de la clase de snoRNA de caja C / D ; sin embargo, a diferencia de otros ARNsno de caja C / D, no se ha demostrado que dirija la metilación 2'- O - de otros ARN. Más bien, se cree que U3 guía la escisión específica del sitio del ARN ribosómico (ARNr) durante el procesamiento del prerARN.
El elemento de caja C / D es un subconjunto de los seis elementos de secuencia corta que se encuentran en todos los snoRNA de U3, a saber, las cajas A, A ', B, C, C' y D. La estructura secundaria de snoRNA de U3 se caracteriza por un pequeño 5 ' dominio (con casillas A y A '), y un dominio 3' más grande (con casillas B, C, C 'y D), estando los dos dominios enlazados por una bisagra monocatenaria. Las cajas B y C forman el motivo B / C, que parece ser exclusivo de los snoRNA de U3, y las cajas C 'y D forman el motivo C' / D. Este último es funcionalmente similar a los motivos C / D que se encuentran en otros snoRNA. El dominio 5 'y la región bisagra actúan como un dominio de unión a pre-rRNA. El dominio 3 'tiene sitios de unión a proteínas conservados. Tanto los motivos de la caja B / C como de la caja C '/ D son suficientes para la retención nuclear del ARNsno U3. El motivo de la caja C '/ D también es necesario para la localización nucleolar, la estabilidad y la hipermetilación del snoRNA U3. Tanto los motivos de caja B / C como C '/ D están involucrados en interacciones proteicas específicas y son necesarios para las funciones de procesamiento del ARNr del ARNsno U3.
Modelos de estructura secundaria específicos de la especie
Está disponible la estructura secundaria de S. cerevisiae determinada por mapeo químico del ARN U3A en un snoRNP purificado. Un humano modelo de estructura también se ha propuesto. Como la levadura y el ser humano, el protista protozoario Entamoeba histolytica: un eucariota primitivo adoptó la misma estructura secundaria conservada del snoRNA U3. Se han propuesto cuatro estructuras de consenso específicas para metazoos , hongos , plantas y eucariotas basales.
Ver también
Referencias
enlaces externos
- Página de ARN nucleolar pequeño U3 en Rfam
- uRNADB: página U3 (archivo)
- La entrada de UMASS snoRNAdb para U3
- La entrada SGD para U3a
- La entrada de snoRNAbase humana para U3
| Este artículo de biología molecular es un fragmento . Puedes ayudar a Wikipedia expandiéndolo . |
