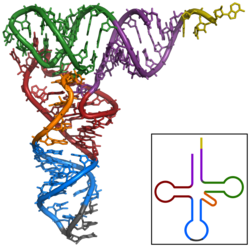
ARN no codificante - Non-coding RNA

Un ARN no codificante ( ncRNA ) es una molécula de ARN que no se traduce en una proteína . La secuencia de ADN a partir de la cual se transcribe un ARN funcional no codificante a menudo se denomina gen de ARN . Abundantes y funcionalmente importantes tipos de ARN no codificantes incluyen ARN de transferencia (ARNt) y ARN ribosómico (ARNr), así como pequeños ARN tales como microARN , ARNip , piRNAs , snoRNAs , ARNsn , exRNAs , scaRNAs y los largos ncRNAs como Xist y HOTAIR .
Se desconoce el número de ARN no codificantes dentro del genoma humano; sin embargo, estudios transcriptómicos y bioinformáticos recientes sugieren que hay miles de ellos. Muchos de los ncRNA recién identificados no han sido validados para su función. También es probable que muchos ncRNA no sean funcionales (a veces denominados ARN basura ) y sean el producto de una transcripción falsa.
Se cree que los ARN no codificantes contribuyen a enfermedades como el cáncer y el Alzheimer .
Historia y descubrimiento
Los ácidos nucleicos fueron descubiertos por primera vez en 1868 por Friedrich Miescher y en 1939 el ARN estaba implicado en la síntesis de proteínas . Dos décadas más tarde, Francis Crick predijo un componente de ARN funcional que mediaba la traducción ; razonó que el ARN se adapta mejor a un par de bases con una transcripción de ARNm que con un polipéptido puro .
El primer ARN no codificante que se caracterizó fue un ARNt de alanina que se encuentra en la levadura de panadería , su estructura se publicó en 1965. Para producir una muestra de ARNt de alanina purificada, Robert W. Holley et al. usó 140 kg de levadura de panadería comercial para dar solo 1 g de ARNt Ala purificado para el análisis. El tRNA de 80 nucleótidos se secuenció primero digeriéndolo con ribonucleasa pancreática (produciendo fragmentos que terminan en citosina o uridina ) y luego con takadiastasa ribonucleasa T1 (produciendo fragmentos que terminaron con guanosina ). La cromatografía y la identificación de los extremos 5 'y 3' ayudaron a organizar los fragmentos para establecer la secuencia de ARN. De las tres estructuras propuestas originalmente para este ARNt, la estructura de 'hoja de trébol' se propuso de forma independiente en varias publicaciones siguientes. La estructura secundaria de la hoja de trébol se finalizó después de un análisis de cristalografía de rayos X realizado por dos grupos de investigación independientes en 1974.
A continuación se descubrió el ARN ribosómico , seguido del URNA a principios de la década de 1980. Desde entonces, el descubrimiento de nuevos ARN no codificantes ha continuado con snoRNA , Xist , CRISPR y muchos más. Las adiciones notables recientes incluyen riboswitches y miARN ; el descubrimiento del mecanismo de ARNi asociado con este último le valió a Craig C. Mello y Andrew Fire el Premio Nobel de Fisiología o Medicina de 2006 .
Los descubrimientos recientes de ncRNA se han logrado mediante métodos tanto experimentales como bioinformáticos .
Roles biológicos
Los ARN no codificantes pertenecen a varios grupos y están involucrados en muchos procesos celulares. Estos van desde ncRNA de importancia central que se conservan en toda o la mayor parte de la vida celular hasta ncRNA más transitorios específicos de una o unas pocas especies estrechamente relacionadas. Se cree que los ncRNA más conservados son fósiles moleculares o reliquias del último ancestro común universal y del mundo del ARN , y sus funciones actuales siguen siendo principalmente en la regulación del flujo de información del ADN a la proteína.
En traducción

Muchos de los ncRNA conservados, esenciales y abundantes están implicados en la traducción . Las partículas de ribonucleoproteína (RNP) llamadas ribosomas son las 'fábricas' donde tiene lugar la traducción en la célula. El ribosoma consta de más del 60% de ARN ribosómico ; estos se componen de 3 ncRNA en procariotas y 4 ncRNA en eucariotas . Los ARN ribosomales catalizan la traducción de secuencias de nucleótidos a proteínas. Otro conjunto de ncRNA, Transfer RNA , forma una "molécula adaptadora" entre el mRNA y la proteína. Los snoRNA de la caja H / ACA y la caja C / D son ncRNA que se encuentran en arqueas y eucariotas. La RNasa MRP está restringida a eucariotas. Ambos grupos de ncRNA están involucrados en la maduración del rRNA. Los snoRNA guían modificaciones covalentes de rRNA, tRNA y snRNA ; La ARNasa MRP escinde el espaciador transcrito interno 1 entre los ARNr 18S y 5.8S. El ubicuo ncRNA, RNase P , es un pariente evolutivo de RNase MRP. La ARNasa P madura las secuencias de ARNt generando extremos 5 'maduros de ARNt mediante la escisión de los elementos líderes en 5' de los ARNt precursores. Otro RNP ubicuo llamado SRP reconoce y transporta proteínas nacientes específicas al retículo endoplásmico en eucariotas y la membrana plasmática en procariotas . En bacterias , el ARN mensajero de transferencia (ARNtm) es un RNP involucrado en rescatar ribosomas estancados, etiquetar polipéptidos incompletos y promover la degradación del ARNm aberrante.
En el empalme de ARN
En eucariotas, el espliceosoma realiza las reacciones de corte y empalme esenciales para eliminar las secuencias de intrones , este proceso es necesario para la formación de ARNm maduro . El espliceosoma es otro RNP a menudo también conocido como snRNP o tri-snRNP. Hay dos formas diferentes del espliceosoma, la forma mayor y la menor. Los componentes del ARNc del principal espliceosoma son U1 , U2 , U4 , U5 y U6 . Los componentes de ncRNA del espliceosoma menor son U11 , U12 , U5 , U4atac y U6atac .
Otro grupo de intrones puede catalizar su propia eliminación de las transcripciones del huésped; estos se denominan ARN autoempalmes. Hay dos grupos principales de ARN auto-empalmados: intrón catalítico del grupo I e intrón catalítico del grupo II . Estos ncRNA catalizan su propia escisión de precursores de mRNA, tRNA y rRNA en una amplia gama de organismos.
En mamíferos, se ha descubierto que los snoRNA también pueden regular el empalme alternativo del mRNA, por ejemplo, el snoRNA HBII-52 regula el empalme del receptor de serotonina 2C .
En los nematodos, el ncRNA SmY parece estar involucrado en el empalme trans de mRNA .
En la replicación del ADN

Los ARN Y son bucles madre, necesarios para la replicación del ADN a través de interacciones con la cromatina y las proteínas de iniciación (incluido el complejo de reconocimiento de origen ). También son componentes de la partícula de ribonucleoproteína Ro60 que es un objetivo de los anticuerpos autoinmunes en pacientes con lupus eritematoso sistémico .
En regulación genética
La expresión de muchos miles de genes está regulada por ncRNA. Esta regulación puede ocurrir en trans o en cis . Existe una creciente evidencia de que un tipo especial de ncRNA llamados ARN potenciadores , transcritos de la región potenciadora de un gen, actúan para promover la expresión génica.
Trans-actuando
En eucariotas superiores, los microARN regulan la expresión génica. Un solo miARN puede reducir los niveles de expresión de cientos de genes. El mecanismo por el cual actúan las moléculas de miARN maduras es mediante complementación parcial de una o más moléculas de ARN mensajero (ARNm), generalmente en 3 'UTR . La función principal de los miARN es regular a la baja la expresión génica.
También se ha demostrado que el ncRNA RNase P influye en la expresión génica. En el núcleo humano, la ARNasa P es necesaria para la transcripción normal y eficaz de varios ARNc transcritos por la ARN polimerasa III . Estos incluyen los genes tRNA, 5S rRNA , SRP RNA y U6 snRNA . La ARNasa P ejerce su papel en la transcripción a través de la asociación con Pol III y cromatina de genes activos de ARNt y ARNr 5S.
Se ha demostrado que el ARN 7SK , un ARNn de metazoos , actúa como regulador negativo del factor de elongación de la ARN polimerasa II P-TEFb , y que esta actividad está influenciada por las vías de respuesta al estrés.
El ncRNA bacteriano, 6S RNA , se asocia específicamente con la holoenzima de la RNA polimerasa que contiene el factor de especificidad sigma70 . Esta interacción reprime la expresión de un promotor dependiente de sigma70 durante la fase estacionaria .
Otro ncRNA bacteriano, OxyS RNA reprime la traducción uniéndose a secuencias de Shine-Dalgarno ocluyendo así la unión de ribosomas. El ARN de OxyS se induce en respuesta al estrés oxidativo en Escherichia coli.
El ARN B2 es un pequeño transcrito de ARN polimerasa III no codificante que reprime la transcripción del ARNm en respuesta al choque térmico en células de ratón. El ARN B2 inhibe la transcripción uniéndose al núcleo Pol II. A través de esta interacción, el ARN B2 se ensambla en complejos de preiniciación en el promotor y bloquea la síntesis de ARN.
Un estudio reciente ha demostrado que solo el acto de transcripción de la secuencia de ncRNA puede influir en la expresión génica. La transcripción de ARN polimerasa II de ncRNA es necesaria para la remodelación de la cromatina en Schizosaccharomyces pombe . La cromatina se convierte progresivamente en una configuración abierta, a medida que se transcriben varias especies de ncRNA.
Cis-actuando
Varios ncRNA están incrustados en las 5 ' UTR (regiones no traducidas) de genes que codifican proteínas e influyen en su expresión de diversas formas. Por ejemplo, un riboswitch puede unirse directamente a una pequeña molécula diana ; la unión del objetivo afecta la actividad del gen.
Las secuencias líder de ARN se encuentran cadena arriba del primer gen de operones biosintéticos de aminoácidos. Estos elementos de ARN forman una de dos posibles estructuras en regiones que codifican secuencias peptídicas muy cortas que son ricas en el aminoácido producto final del operón. Se forma una estructura de terminación cuando hay un exceso del aminoácido regulador y no se impide el movimiento del ribosoma sobre la transcripción líder. Cuando hay una deficiencia del ARNt cargado del aminoácido regulador, el ribosoma que traduce el péptido líder se detiene y se forma la estructura antiterminador. Esto permite que la ARN polimerasa transcriba el operón. Los líderes de ARN conocidos son el líder del operón de histidina , el líder del operón de leucina , el líder del operón de treonina y el líder del operón de triptófano .
Los elementos de respuesta al hierro (IRE) están unidos por proteínas de respuesta al hierro (IRP). El IRE se encuentra en UTR de varios ARNm cuyos productos están involucrados en el metabolismo del hierro . Cuando la concentración de hierro es baja, los IRP se unen al ARNm de ferritina IRE, lo que conduce a la represión de la traducción.
Los sitios de entrada de ribosomas internos (IRES) son estructuras de ARN que permiten el inicio de la traducción en medio de una secuencia de ARNm como parte del proceso de síntesis de proteínas .
En defensa del genoma
Los ARN que interactúan con Piwi (piRNA) expresados en testículos de mamíferos y células somáticas forman complejos ARN-proteína con proteínas Piwi . Estos complejos piRNA (piRC) se han relacionado con el silenciamiento génico transcripcional de retrotransposones y otros elementos genéticos en las células de la línea germinal , en particular las de la espermatogénesis .
Las repeticiones palindrómicas cortas agrupadas regularmente interespaciadas (CRISPR) son repeticiones que se encuentran en el ADN de muchas bacterias y arqueas . Las repeticiones están separadas por espaciadores de longitud similar. Se ha demostrado que estos espaciadores pueden derivarse de fagos y, posteriormente, ayudar a proteger la célula de la infección.
Estructura cromosómica
La telomerasa es una enzima RNP que agrega repeticiones de secuencias de ADN específicas ("TTAGGG" en los vertebrados) a las regiones teloméricas , que se encuentran en los extremos de los cromosomas eucariotas . Los telómeros contienen material de ADN condensado, lo que da estabilidad a los cromosomas. La enzima es una transcriptasa inversa que transporta ARN de telomerasa , que se utiliza como plantilla cuando alarga los telómeros, que se acortan después de cada ciclo de replicación .
Xist (transcripción específica de X-inactivo) es un gen ncRNA largo en el cromosoma X de los mamíferos placentarios que actúa como efector principal del proceso de inactivación del cromosoma X que forma los cuerpos de Barr . Un ARN antisentido , Tsix , es un regulador negativo de Xist. Los cromosomas X que carecen de expresión de Tsix (y por lo tanto tienen altos niveles de transcripción de Xist) se inactivan con más frecuencia que los cromosomas normales. En las drosofílidas , que también utilizan un sistema de determinación del sexo XY , los ARN roX (ARN en el X) participan en la compensación de la dosis. Tanto Xist como roX operan mediante la regulación epigenética de la transcripción mediante el reclutamiento de enzimas modificadoras de histonas .
ARN bifuncional
Los ARN bifuncionales , o ARN de función dual , son ARN que tienen dos funciones distintas. La mayoría de los ARN bifuncionales conocidos son ARNm que codifican tanto una proteína como ncRNA. Sin embargo, un número creciente de ncRNA se clasifican en dos categorías diferentes de ncRNA; por ejemplo, ARNsno y miARN de caja H / ACA .
Dos ejemplos bien conocidos de ARN bifuncionales son ARN SgrS y ARNIII . Sin embargo, se sabe que existen algunos otros ARN bifuncionales (p. Ej., Activador del receptor de esteroides / SRA, ARN VegT, ARN Oskar, ENOD40 , ARN p53 y ARN SR1 . Los ARN bifuncionales han sido recientemente el tema de un número especial de Biochimie .
Como una hormona
Existe un vínculo importante entre ciertos ARN no codificantes y el control de las vías reguladas por hormonas. En Drosophila , hormonas como la ecdisona y la hormona juvenil pueden promover la expresión de ciertos miARN. Además, esta regulación se produce en distintos puntos temporales dentro del desarrollo de C. elegans . En los mamíferos, el miR-206 es un regulador crucial del receptor alfa de estrógenos .
Los ARN no codificantes son cruciales en el desarrollo de varios órganos endocrinos, así como en enfermedades endocrinas como la diabetes mellitus . Específicamente en la línea celular MCF-7, la adición de 17β- estradiol aumentó la transcripción global de los ARN no codificantes llamados lncRNA cerca de los genes codificantes activados por estrógenos.
En evitación patógena
Se demostró que C. elegans aprende y hereda la evitación patógena después de la exposición a un solo ARN no codificante de un patógeno bacteriano .
Funciones en la enfermedad
Al igual que ocurre con las proteínas , las mutaciones o los desequilibrios en el repertorio de ncRNA dentro del cuerpo pueden causar una variedad de enfermedades.
Cáncer
Muchos ncRNA muestran patrones de expresión anormales en tejidos cancerosos . Estos incluyen miRNAs , largo mRNA-como ncRNAs , GAS5 , SNORD50 , telomerasa ARN y Y ARNs . Los miRNA están involucrados en la regulación a gran escala de muchos genes codificadores de proteínas, los Y RNA son importantes para el inicio de la replicación del ADN, el ARN de la telomerasa que sirve como cebador para la telomerasa, un RNP que extiende las regiones teloméricas en los extremos de los cromosomas (ver telómeros y enfermedad para obtener más información). La función directa de los ncRNA similares a ARNm largos es menos clara.
Se ha demostrado que las mutaciones de la línea germinal en los precursores primarios de miR-16-1 y miR-15 son mucho más frecuentes en pacientes con leucemia linfocítica crónica en comparación con las poblaciones de control.
Se ha sugerido que un SNP poco común ( rs11614913 ) que se superpone a has-mir-196a2 se ha asociado con el carcinoma de pulmón de células no pequeñas . Asimismo, un cribado de 17 miARN que se ha predicho que regulan una serie de genes asociados al cáncer de mama encontró variaciones en los microARN miR-17 y miR-30c-1 de las pacientes; estos pacientes no eran portadores de mutaciones BRCA1 o BRCA2 , lo que abre la posibilidad de que el cáncer de mama familiar pueda ser causado por la variación en estos miARN. El supresor de tumores p53 es posiblemente el agente más importante para prevenir la formación y progresión de tumores. La proteína p53 funciona como un factor de transcripción con un papel crucial en la orquestación de la respuesta al estrés celular. Además de su papel crucial en el cáncer, p53 se ha implicado en otras enfermedades como diabetes, muerte celular después de isquemia y diversas enfermedades neurodegenerativas como Huntington, Parkinson y Alzheimer. Los estudios han sugerido que la expresión de p53 está sujeta a regulación por ARN no codificante.
Otro ejemplo de ARN no codificante desregulado en células cancerosas es el ARN no codificante largo Linc00707. Linc00707 está regulado positivamente y esponja miARN en células madre mesenquimatosas derivadas de la médula ósea humana, en carcinoma hepatocelular, cáncer gástrico o cáncer de mama, y así promueve la osteogénesis, contribuye a la progresión del carcinoma hepatocelular, promueve la proliferación y metástasis, o regula indirectamente la expresión de proteínas involucradas en agresividad del cáncer, respectivamente.
Síndrome de Prader-Willi
Se ha demostrado que la supresión de las 48 copias de la caja C / D snoRNA SNORD116 es la causa principal del síndrome de Prader-Willi . Prader-Willi es un trastorno del desarrollo asociado con la sobrealimentación y las dificultades de aprendizaje. SNORD116 tiene sitios diana potenciales dentro de varios genes que codifican proteínas y podría tener un papel en la regulación del corte y empalme alternativo.
Autismo
El locus cromosómico que contiene el pequeño grupo de genes de ARN nucleolar SNORD115 se ha duplicado en aproximadamente el 5% de los individuos con rasgos autistas . Un modelo de ratón diseñado para tener una duplicación del grupo SNORD115 muestra un comportamiento similar al autista. Un pequeño estudio reciente de tejido cerebral post-mortem demostró una expresión alterada de ARN largos no codificantes en la corteza prefrontal y el cerebelo de cerebros autistas en comparación con los controles.
Hipoplasia cartílago-cabello
Se ha demostrado que las mutaciones dentro de la RNasa MRP causan hipoplasia del cartílago y el cabello , una enfermedad asociada con una variedad de síntomas como baja estatura, cabello escaso, anomalías esqueléticas y un sistema inmunológico debilitado que es frecuente entre los amish y los finlandeses . La variante mejor caracterizada es una transición A-a-G en el nucleótido 70 que está en una región de bucle dos bases 5 'de un pseudonudo conservado . Sin embargo, muchas otras mutaciones dentro de la ARNasa MRP también causan CHH.
Enfermedad de Alzheimer
El ARN antisentido, BACE1-AS se transcribe desde la hebra opuesta a BACE1 y se regula al alza en pacientes con enfermedad de Alzheimer . BACE1-AS regula la expresión de BACE1 aumentando la estabilidad del ARNm de BACE1 y generando BACE1 adicional a través de un mecanismo de retroalimentación postranscripcional. Por el mismo mecanismo también aumenta las concentraciones de beta amiloide , el principal componente de las placas seniles. Las concentraciones de BACE1-AS están elevadas en sujetos con enfermedad de Alzheimer y en ratones transgénicos de proteína precursora amiloide.
miR-96 y pérdida auditiva
La variación dentro de la región de la semilla del miR-96 maduro se ha asociado con una pérdida auditiva progresiva autosómica dominante en humanos y ratones. Los ratones mutantes homocigotos eran profundamente sordos y no mostraban respuestas cocleares . Los ratones heterocigotos y los humanos pierden progresivamente la capacidad de oír.
Distinción entre ARN funcional (ARNf) y ARNnc
Los científicos han comenzado a distinguir el ARN funcional ( ARNf ) del ARNnc , para describir regiones funcionales a nivel de ARN que pueden o no ser transcripciones de ARN independientes. Esto implica que el ARNf (como los riboswitches, los elementos SECIS y otras regiones reguladoras en cis) no es ARNc. Sin embargo, el ARNf también podría incluir ARNm , ya que es ARN que codifica proteínas y, por lo tanto, es funcional. Además, los ARN evolucionados artificialmente también se incluyen en el término genérico de ARNf. Algunas publicaciones afirman que ncRNA y fRNA son casi sinónimos, sin embargo, otras han señalado que una gran proporción de ncRNA anotados probablemente no tienen función. También se ha sugerido usar simplemente el término ARN , ya que la distinción de un ARN codificante de proteína ( ARN mensajero ) ya está dada por el calificador ARNm . Esto elimina la ambigüedad al abordar un gen que "codifica un ARN no codificante". Además, puede haber una serie de ncRNA que se mencionan incorrectamente en la literatura y los conjuntos de datos publicados.
Ver también
- ARN extracelular
- Lista de ARN
- Estructura del ácido nucleico
- Rfam
- Riboswitch
- Ribozima
- ARN presentes en muestras ambientales
- ARN VA (asociado a virus)
Referencias
enlaces externos
( Copia de Wayback Machine )
- La base de datos Rfam : una lista seleccionada de cientos de familias de ncRNA relacionados
- NONCODE.org : una base de datos gratuita de todo tipo de ARN no codificantes (excepto ARNt y ARNr)
- RNAcon Predicción y clasificación de ncRNA BMC Genomics 2014, 15: 127
- Explorador de subprocesos ENCODE Caracterización de ARN no codificante. Naturaleza (diario)
- El recurso de bases de datos de ARN no codificante (NRDR) : una fuente seleccionada de datos relacionados con bases de datos de ARN no codificantes disponibles en Internet.
- DASHR : una base de datos de pequeños ARN no codificantes Bioinformática 2018