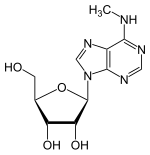
N 6- Metiladenosina - N6-Methyladenosine
|
|
| Nombres | |
|---|---|
|
Nombre IUPAC
N- metiladenosina
|
|
|
Nombre IUPAC preferido
(2 R , 3 S , 4 R , 5 R ) -2- (hidroximetil) -5- [6- (metilamino) -9 H -purin-9-il] oxolano-2,3-diol |
|
| Otros nombres
m 6 A
|
|
| Identificadores | |
|
Modelo 3D ( JSmol )
|
|
| CHEBI | |
| ChemSpider | |
|
PubChem CID
|
|
| UNII | |
|
Tablero CompTox ( EPA )
|
|
|
|
|
|
| Propiedades | |
| C 11 H 15 N 5 O 4 | |
| Masa molar | 281,272 g · mol −1 |
|
Salvo que se indique lo contrario, los datos se proporcionan para materiales en su estado estándar (a 25 ° C [77 ° F], 100 kPa). |
|
| Referencias de Infobox | |
La N 6- metiladenosina ( m 6 A ) se identificó originalmente y se caracterizó parcialmente en la década de 1970, y es una modificación abundante en el ARNm y el ADN. Se encuentra en algunos virus y en la mayoría de los eucariotas, incluidos mamíferos, insectos, plantas y levaduras. También se encuentra en tRNA , rRNA y RNA nuclear pequeño (snRNA), así como en varios RNA largos no codificantes , como Xist .
La metilación de la adenosina está dirigida por un gran m 6 Un metiltransferasa complejo que contiene METTL3 como la sub-unidad SAM vinculante. In vitro , este complejo de metiltransferasa metila preferentemente oligonucleótidos de ARN que contienen GGACU y se identificó una preferencia similar in vivo en sitios m 6 A mapeados en ARN genómico del virus del sarcoma de Rous y en ARNm de prolactina bovina. Estudios más recientes han caracterizado otros componentes clave del complejo de metiltransferasa m 6 A en mamíferos, incluyendo METTL14 , proteína asociada al tumor de Wilms 1 (WTAP), KIAA1429 y METTL5. Tras una especulación de 2010 de que m 6 A en el ARNm es dinámico y reversible, el descubrimiento de la primera m 6 A desmetilasa, masa grasa y proteína asociada a la obesidad (FTO) en 2011 confirmó esta hipótesis y revitalizó el interés en el estudio de m 6 A. Una segunda m 6 Un desmetilasa AlkB homólogo 5 (ALKBH5) más tarde se descubrió también.
Las funciones biológicas de m 6 A están mediadas por un grupo de proteínas de unión a ARN que reconocen específicamente la adenosina metilada en el ARN. Estas proteínas de unión se nombran m 6 lectores a. La familia de proteínas del dominio de homología YT521-B (YTH) ( YTHDF1 , YTHDF2 , YTHDF3 e YTHDC1 ) se ha caracterizado como lectores directos m 6 A y tiene un bolsillo de unión m 6 A conservado . Las proteínas 1, 2 y 3 que se unen al ARNm del factor de crecimiento similar a la insulina 2 (IGF2BP1–3) se describen como una nueva clase de lectores de m6A. Las IGF2BP usan dominios de homología K (KH) para reconocer selectivamente ARN que contienen m6A y promover su traducción y estabilidad. Estos lectores de m 6 A, junto con las metiltransferasas de m 6 A (escritores) y demetilasas (borradores), establecen un mecanismo complejo de regulación de m 6 A en el que los escritores y borradores determinan las distribuciones de m 6 A en el ARN, mientras que los lectores median m 6 A Funciones dependientes de A. También se ha demostrado que m 6 A media en un interruptor estructural denominado interruptor m 6 A.
Distribución de especies
Levadura
En la levadura en ciernes ( Sacharomyces cerevisiae ), el homólogo de METTL3 , IME4 se induce en células diploides en respuesta a la inanición de la fuente de nitrógeno y carbono fermentable y es necesario para la metilación del ARNm y el inicio de la meiosis y la esporulación correctas. Se sabe que los ARNm de IME1 e IME2, reguladores tempranos clave de la meiosis , son objetivos para la metilación , al igual que las transcripciones del propio IME4.
Plantas
En las plantas, la mayoría de m 6 A se encuentra dentro de los 150 nucleótidos antes del comienzo de la cola de poli (A) .
Las mutaciones de MTA, el homólogo de Arabidopsis thaliana de METTL3, dan como resultado la detención del embrión en la etapa globular. Una reducción> 90% de los niveles de m 6 A en plantas maduras conduce a patrones de crecimiento dramáticamente alterados y anomalías homeóticas florales.
Mamíferos
El mapeo de m 6 A en ARN humano y de ratón ha identificado más de 18 000 sitios m 6 A en las transcripciones de más de 7 000 genes humanos con una secuencia de consenso de [G / A / U] [G> A] m 6 AC [U> A / C] coherente con el motivo previamente identificado. La localización de sitios m 6 A individuales en muchos ARNm es muy similar entre humanos y ratones , y el análisis de todo el transcriptoma revela que m 6 A se encuentra en regiones de alta conservación evolutiva . m 6 A se encuentra dentro de los exones internos largos y se enriquece preferentemente dentro de las UTR 3 'y alrededor de los codones de terminación . m 6 A dentro de 3 'UTR también se asocia con la presencia de sitios de unión de microARN; más o menos 2/3 de los ARNm que contienen un m 6 Un sitio dentro de su 3' UTR también tienen al menos un microRNA sitio de unión. Al integrar todos los datos de secuenciación de m 6 A, una nueva base de datos llamada RMBase ha identificado y proporcionado aproximadamente 200.000 sitios en los genomas humanos y de ratón correspondientes a N6-metiladenosinas (m 6 A) en el ARN.
El mapeo preciso de m6A por m6A-CLIP / IP (brevemente m6A- CLIP ) reveló que la mayoría de m6A se localiza en el último exón de ARNm en múltiples tejidos / células cultivadas de ratón y humano, y el enriquecimiento de m6A alrededor de los codones de parada es una coincidencia que muchos codones de parada se ubican alrededor del comienzo de los últimos exones donde m6A está verdaderamente enriquecido. La mayor presencia de m6A en el último exón (> = 70%) permite el potencial de regulación de 3'UTR, incluida la poliadenilación alternativa. El estudio que combina m6A-CLIP con una rigurosa bioquímica de fraccionamiento celular revela que las modificaciones del mRNA de m6A se depositan en el pre-mRNA naciente y no son necesarias para el empalme, pero especifican el recambio citoplásmico.
m 6 A es susceptible de regulación dinámica tanto durante el desarrollo como en respuesta a estímulos celulares. El análisis de m 6 A en el ARN de cerebro de ratón revela que los niveles de m 6 A son bajos durante el desarrollo embrionario y aumentan drásticamente en la edad adulta. Además, silenciar la metiltransferasa m 6 A afecta significativamente la expresión génica y los patrones de empalme de ARN alternativos , lo que resulta en la modulación de la vía de señalización p53 (también conocida como TP53 ) y la apoptosis .
El m 6 A también se encuentra en los componentes de ARN de los bucles R en las células humanas, donde participa en la regulación de la estabilidad de los híbridos ARN: ADN.
Recientemente se demostró la importancia de la metilación de m 6 A para los procesos fisiológicos. La inhibición de la m 6 Un metilación a través de la inhibición farmacológica de metilaciones celulares o más específicamente por el silenciamiento mediado por ARNsi de la m 6 Un metilasa Mettl3 llevó a la elongación de la circadiano período. Por el contrario, la sobreexpresión de Mettl3 condujo a un período más corto. El reloj circadiano de los mamíferos , compuesto por un bucle de retroalimentación de transcripción estrictamente regulado para oscilar con un período de aproximadamente 24 horas, es por lo tanto extremadamente sensible a las perturbaciones en el procesamiento del ARN dependiente de m 6 A, probablemente debido a la presencia de sitios m 6 A dentro del reloj. transcripciones de genes. Los efectos de la inhibición de la metilación global sobre el período circadiano en células de ratón se pueden prevenir mediante la expresión ectópica de una enzima del metabolismo del metilo bacteriano. Las células de ratón que expresan esta proteína bacteriana fueron resistentes a la inhibición farmacológica del metabolismo del metilo, y no mostraron disminución en la metilación de mRNA m 6 A o metilación de proteínas .
Significación clínica
Considerando las funciones versátiles de m 6 A en varios procesos fisiológicos, no es sorprendente encontrar vínculos entre m 6 A y numerosas enfermedades humanas; muchos se originaron a partir de mutaciones o polimorfismos de un solo nucleótido (SNP) de factores afines de m 6 A. Los vínculos entre m 6 A y numerosos tipos de cáncer se han indicado en informes que incluyen cáncer de estómago, cáncer de próstata, cáncer de mama, cáncer de páncreas, cáncer de riñón , mesotelioma, sarcoma y leucemia. Los impactos de m 6 A en la proliferación de células cancerosas podrían ser mucho más profundos con la aparición de más datos. Se sabe que el agotamiento de METTL3 causa la apoptosis de las células cancerosas y reduce la invasividad de las células cancerosas, mientras que se demostró que la activación de ALKBH5 por hipoxia causa el enriquecimiento de las células madre cancerosas. El m 6 A también se ha indicado en la regulación de la homeostasis energética y la obesidad, ya que FTO es un gen regulador clave para el metabolismo energético y la obesidad. Se ha demostrado que los SNP de FTO se asocian con el índice de masa corporal en poblaciones humanas y la aparición de obesidad y diabetes. Se ha sugerido la influencia de FTO en la diferenciación de preadipocitos. También se ha estudiado la conexión entre m 6 A y trastornos neuronales. Por ejemplo, las enfermedades neurodegenerativas pueden verse afectadas por m 6 A ya que se demostró que la señalización de la dopamina análoga depende de FTO y corrige la metilación de m 6 A en las transcripciones de señalización clave. Se sabe que las mutaciones en HNRNPA2B1, un lector potencial de m 6 A, causan neurodegeneración. El IGF2BP1–3, una nueva clase de lector m 6 A, tiene funciones oncogénicas. La desactivación o desactivación de IGF2BP1–3 disminuyó la expresión de la proteína MYC, la proliferación celular y la formación de colonias en líneas celulares de cáncer humano. El ZC3H13, un miembro del complejo de metiltransferasa m6A, inhibió notablemente el crecimiento de las células de cáncer colorrectal cuando se derribó.
Además, se ha informado que m 6 A afecta las infecciones virales. Se sabe que muchos virus de ARN, incluidos SV40, adenovirus, virus del herpes, virus del sarcoma de Rous y virus de la influenza, contienen metilación interna de m 6 A en el ARN genómico del virus. Varios estudios más recientes han revelado que los reguladores de m 6 A gobiernan la eficiencia de la infección y la replicación de virus de ARN como el virus de la inmunodeficiencia humana (VIH), el virus de la hepatitis C (VHC) y el virus del Zika (ZIKV). Estos resultados sugieren que m 6 A y sus factores afines juegan un papel crucial en la regulación de los ciclos de vida del virus y las interacciones entre el huésped y el virus.
