microARN - microRNA
Un microARN ( miARN abreviado ) es una pequeña molécula de ARN no codificante monocatenario (que contiene aproximadamente 22 nucleótidos ) que se encuentra en plantas, animales y algunos virus, que funciona en el silenciamiento del ARN y la regulación postranscripcional de la expresión génica . Los miARN funcionan a través del emparejamiento de bases con secuencias complementarias dentro de las moléculas de ARNm . Como resultado, estas moléculas de ARNm se silencian mediante uno o más de los siguientes procesos: (1) escisión de la hebra de ARNm en dos partes, (2) desestabilización del ARNm mediante el acortamiento de su cola poli (A) y ( 3) traducción menos eficiente del ARNm en proteínas por los ribosomas .
Los miARN se asemejan a los pequeños ARN interferentes (ARNip) de la vía del ARN de interferencia (ARNi) , excepto que los miARN se derivan de regiones de transcripciones de ARN que se pliegan sobre sí mismas para formar horquillas cortas, mientras que los ARNsi se derivan de regiones más largas de ARN bicatenario . El genoma humano puede codificar más de 1900 miARN, aunque un análisis más reciente sugiere que el número se acerca a los 2300. Sin embargo, solo alrededor de 500 microARN humanos representan miARN genuino en la base de datos de genes de miARN curada manualmente MirGeneDB .
Los miARN son abundantes en muchos tipos de células de mamíferos y como miARN circulantes extracelulares . Los miARN circulantes se liberan en los fluidos corporales, incluida la sangre y el líquido cefalorraquídeo, y tienen el potencial de estar disponibles como biomarcadores en varias enfermedades. Los miARN parecen dirigirse a aproximadamente el 60% de los genes de los seres humanos y otros mamíferos. Muchos miARN se conservan evolutivamente, lo que implica que tienen funciones biológicas importantes. Por ejemplo, se han conservado 90 familias de miARN desde al menos el ancestro común de mamíferos y peces, y la mayoría de estos miARN conservados tienen funciones importantes, como lo demuestran estudios en los que se han eliminado genes de uno o más miembros de una familia. en ratones.
Historia
El primer miARN se descubrió a principios de la década de 1990. Sin embargo, los miARN no fueron reconocidos como una clase distinta de reguladores biológicos hasta principios de la década de 2000. La investigación de miARN reveló diferentes conjuntos de miARN expresados en diferentes tipos de células y tejidos y múltiples roles para los miARN en el desarrollo de plantas y animales y en muchos otros procesos biológicos. La expresión aberrante de miARN está implicada en estados patológicos. Se están investigando terapias basadas en miARN.
El primer miARN fue descubierto en 1993 por un grupo liderado por Ambros y que incluía a Lee y Feinbaum. Sin embargo, la comprensión adicional de su modo de acción requirió trabajos publicados simultáneamente por el equipo de Ruvkun , incluidos Wightman y Ha. Estos grupos publicaron artículos consecutivos sobre el gen lin-4 , que se sabía que controlaba el tiempo del desarrollo larvario de C. elegans reprimiendo el gen lin-14 . Cuando Lee et al. aislaron el miARN lin-4 , encontraron que en lugar de producir un ARNm que codifica una proteína, producía ARN cortos no codificantes , uno de los cuales era un ARN de ~ 22 nucleótidos que contenía secuencias parcialmente complementarias a múltiples secuencias en el 3 'UTR del ARNm lin-14 . Se propuso que esta complementariedad inhibiera la traducción del ARNm de lin-14 en la proteína LIN-14. En ese momento, se pensaba que el ARN pequeño lin-4 era una idiosincrasia de nematodos .
En 2000, se caracterizó un segundo ARN pequeño: let-7 ARN, que reprime lin-41 para promover una transición de desarrollo posterior en C. elegans . Se encontró que el ARN let-7 se conservaba en muchas especies, lo que llevó a la sugerencia de que el ARN let-7 y los "ARN temporales pequeños" adicionales podrían regular el tiempo de desarrollo en diversos animales, incluidos los humanos.
Un año más tarde, se descubrió que los ARN lin-4 y let-7 eran parte de una gran clase de ARN pequeños presentes en C. elegans , Drosophila y células humanas. Los muchos ARN de esta clase se parecían a los ARN lin-4 y let-7 , excepto que sus patrones de expresión eran generalmente incompatibles con un papel en la regulación del tiempo de desarrollo. Esto sugirió que la mayoría podría funcionar en otros tipos de vías reguladoras. En este punto, los investigadores comenzaron a usar el término "microARN" para referirse a esta clase de pequeños ARN reguladores.
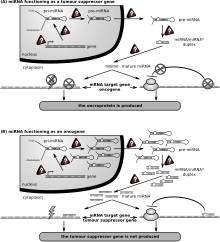
La primera enfermedad humana asociada con la desregulación de los miARN fue la leucemia linfocítica crónica . En este trastorno, los miARN tienen una función dual, actuando como supresores de tumores y como oncogenes.
Nomenclatura
Bajo un sistema de nomenclatura estándar, los nombres se asignan a los miARN confirmados experimentalmente antes de su publicación. El prefijo "miR" va seguido de un guión y un número, este último a menudo indica el orden de denominación. Por ejemplo, miR-124 fue nombrado y probablemente descubierto antes que miR-456. Un "miR-" en mayúsculas se refiere a la forma madura del miRNA, mientras que "mir-" sin mayúsculas se refiere al pre-miRNA y al pri-miRNA. Los miARN que codifican genes también se nombran usando el mismo prefijo de tres letras de acuerdo con las convenciones de la nomenclatura de genes del organismo. Por ejemplo, los nombres de genes oficiales de miARN en algunos organismos son " mir-1 en C. elegans y Drosophila, Mir-1 en Rattus norvegicus y MIR-25 en humanos.
Los miARN con secuencias casi idénticas a excepción de uno o dos nucleótidos se anotan con una letra minúscula adicional. Por ejemplo, miR-124a está estrechamente relacionado con miR-124b. Por ejemplo:
- hsa-miR-181a :aacauucaACgcugucggugAgu
- hsa-miR-181b :aacauucaUUgcugucggugGgu
Los pre-miARN, pri-miARN y genes que conducen a miARN maduros 100% idénticos pero que están ubicados en diferentes lugares del genoma se indican con un sufijo adicional de número de guiones. Por ejemplo, los pre-miARN hsa-mir-194-1 y hsa-mir-194-2 conducen a un miARN maduro idéntico (hsa-miR-194) pero son de genes ubicados en diferentes regiones del genoma.
La especie de origen se designa con un prefijo de tres letras, por ejemplo, hsa-miR-124 es un miARN humano ( Homo sapiens ) y oar-miR-124 es un miARN de oveja ( Ovis aries ). Otros prefijos comunes incluyen "v" para viral (miARN codificado por un genoma viral) y "d" para miARN de Drosophila (una mosca de la fruta comúnmente estudiada en la investigación genética).
Cuando dos microARN maduros se originan a partir de brazos opuestos del mismo pre-miARN y se encuentran en cantidades aproximadamente similares, se indican con un sufijo -3p o -5p. (En el pasado, esta distinción también se hacía con "s" ( sentido ) y "as" (antisentido)). Sin embargo, el microARN maduro que se encuentra en un brazo de la horquilla suele ser mucho más abundante que el que se encuentra en el otro brazo, en cuyo caso, un asterisco después del nombre indica la especie madura que se encuentra en niveles bajos en el brazo opuesto de una horquilla. Por ejemplo, miR-124 y miR-124 * comparten una horquilla pre-miRNA, pero se encuentra mucho más miR-124 en la célula.
Objetivos
Los miARN de plantas suelen tener un apareamiento casi perfecto con sus dianas de ARNm, lo que induce la represión génica a través de la escisión de las transcripciones diana. Por el contrario, los miRNA animales pueden reconocer sus mRNA diana utilizando tan solo 6-8 nucleótidos (la región semilla) en el extremo 5 'del miRNA, que no es suficiente emparejamiento para inducir la escisión de los mRNA diana. La regulación combinatoria es una característica de la regulación de miARN en animales. Un miARN dado puede tener cientos de objetivos de ARNm diferentes, y un objetivo determinado puede estar regulado por múltiples miARN.
Las estimaciones del número promedio de ARN mensajeros únicos que son objetivos de la represión por un miARN típico varían, dependiendo del método de estimación, pero múltiples enfoques muestran que los miARN de mamíferos pueden tener muchos objetivos únicos. Por ejemplo, un análisis de los miARN altamente conservados en vertebrados muestra que cada uno tiene, en promedio, aproximadamente 400 dianas conservadas. Asimismo, los experimentos muestran que una sola especie de miARN puede reducir la estabilidad de cientos de ARN mensajeros únicos. Otros experimentos muestran que una sola especie de miARN puede reprimir la producción de cientos de proteínas, pero que esta represión a menudo es relativamente leve (mucho menos del doble). La primera enfermedad humana que se descubrió que estaba asociada con la desregulación de los miARN fue la leucemia linfocítica crónica . Le siguieron otras neoplasias malignas de células B.
Biogénesis
Hasta el 40% de los genes de miARN pueden estar en los intrones o incluso en los exones de otros genes. Por lo general, aunque no exclusivamente, se encuentran en una orientación de sentido y, por lo tanto, generalmente se regulan junto con los genes del huésped.
La plantilla de ADN no es la última palabra sobre la producción de miARN maduro: el 6% de los miARN humanos muestran edición de ARN ( IsomiR ), la modificación específica del sitio de las secuencias de ARN para producir productos diferentes de los codificados por su ADN. Esto aumenta la diversidad y el alcance de la acción de los miARN más allá de la implicada por el genoma solo.
Transcripción
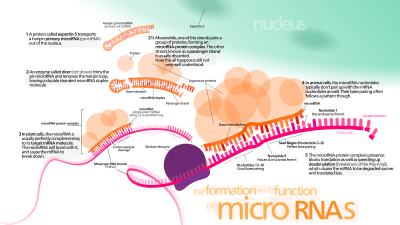
Los genes de miARN generalmente son transcritos por la ARN polimerasa II (Pol II). La polimerasa a menudo se une a un promotor que se encuentra cerca de la secuencia de ADN, codificando lo que se convertirá en el bucle de horquilla del pre-miARN. La transcripción resultante se remata con un nucleótido especialmente modificado en el extremo 5 ', se poliadenila con múltiples adenosinas (una cola poli (A)) y se empalma . Los miARN animales se transcriben inicialmente como parte de un brazo de un tallo-bucle de ARN de ~ 80 nucleótidos que a su vez forma parte de un precursor de miARN de varios cientos de nucleótidos de longitud denominado pri-miARN. Cuando se encuentra un precursor de tallo-bucle en el 3 'UTR, una transcripción puede servir como un pri-miRNA y un mRNA. La ARN polimerasa III (Pol III) transcribe algunos miARN, especialmente aquellos con secuencias de Alu cadena arriba , ARN de transferencia (ARNt) y unidades promotoras de repetición intercalada amplia de mamíferos (MWIR).
Procesamiento nuclear

Un solo pri-miRNA puede contener de uno a seis precursores de miRNA. Estas estructuras de bucles en horquilla están compuestas por aproximadamente 70 nucleótidos cada una. Cada horquilla está flanqueada por secuencias necesarias para un procesamiento eficiente.
La estructura del ARN bicatenario (ARNdc) de las horquillas en un pri-miARN es reconocida por una proteína nuclear conocida como Región crítica del síndrome de DiGeorge 8 (DGCR8 o "Pasha" en invertebrados), llamada así por su asociación con el síndrome de DiGeorge . DGCR8 se asocia con la enzima Drosha , una proteína que corta el ARN, para formar el complejo Microprocesador . En este complejo, DGCR8 orienta el dominio catalítico de ARNasa III de Drosha para liberar horquillas de pri-miARN escindiendo el ARN alrededor de once nucleótidos de la base de la horquilla (un ARNdc helicoidal se convierte en el tallo). El producto resultante tiene un saliente de dos nucleótidos en su extremo 3 '; tiene grupos 3 'hidroxilo y 5' fosfato. A menudo se denomina pre-miARN (precursor-miARN). Se han identificado motivos de secuencia aguas abajo del pre-miARN que son importantes para un procesamiento eficiente.
Los pre-miARN que se empalman directamente de los intrones, sin pasar por el complejo del microprocesador, se conocen como " Mirtron ". Originalmente se pensó que existían solo en Drosophila y C. elegans , ahora se han encontrado mirtrones en mamíferos.
Hasta el 16% de los pre-miARN pueden modificarse mediante la edición de ARN nuclear . Más comúnmente, las enzimas conocidas como adenosina desaminasas que actúan sobre el ARN (ADAR) catalizan las transiciones de adenosina a inosina (A a I). La edición de ARN puede detener el procesamiento nuclear (por ejemplo, de pri-miR-142, que conduce a la degradación por la ribonucleasa Tudor-SN) y alterar los procesos posteriores, incluido el procesamiento de miARN citoplásmico y la especificidad de la diana (por ejemplo, al cambiar la región semilla de miR-376 en el sistema nervioso central).
Exportación nuclear

Las horquillas de pre-miARN se exportan desde el núcleo en un proceso que involucra al transportador nucleocitoplasmático Exportin -5 . Esta proteína, un miembro de la familia de las carioferinas , reconoce un saliente de dos nucleótidos dejado por la enzima RNasa III Drosha en el extremo 3 'de la horquilla pre-miARN. El transporte mediado por exportina-5 al citoplasma depende de la energía, utilizando trifosfato de guanosina (GTP) unido a la proteína Ran .
Procesamiento citoplásmico
En el citoplasma , la horquilla de pre-miARN es escindida por la enzima Dicer de RNasa III . Esta endoribonucleasa interactúa con los extremos 5 'y 3' de la horquilla y corta el lazo que une los brazos 3 'y 5', produciendo un miARN: miARN * dúplex imperfecto de aproximadamente 22 nucleótidos de longitud. La longitud total de la horquilla y el tamaño del bucle influyen en la eficiencia del procesamiento de Dicer. La naturaleza imperfecta del emparejamiento miARN: miARN * también afecta la escisión. Algunos de los pre-miRNA ricos en G pueden adoptar potencialmente la estructura G-quadruplex como una alternativa a la estructura canónica de tallo-bucle. Por ejemplo, el pre-miRNA 92b humano adopta una estructura G-quadruplex que es resistente a la escisión mediada por Dicer en el citoplasma . Aunque cualquiera de las cadenas del dúplex puede actuar potencialmente como un miARN funcional, solo una cadena se incorpora generalmente en el complejo de silenciamiento inducido por ARN (RISC) donde interactúan el miARN y su ARNm diana.
Si bien la mayoría de los miARN se encuentran dentro de la célula, algunos miARN, comúnmente conocidos como miARN circulantes o miARN extracelulares, también se han encontrado en el ambiente extracelular, incluidos varios fluidos biológicos y medios de cultivo celular.
Biogénesis en plantas
La biogénesis de miARN en plantas se diferencia de la biogénesis animal principalmente en los pasos de procesamiento nuclear y exportación. En lugar de ser escindidos por dos enzimas diferentes, una vez dentro y fuera del núcleo, ambas escisiones del miARN de la planta son realizadas por un homólogo de Dicer, llamado Dicer-like1 (DL1). DL1 se expresa solo en el núcleo de las células vegetales, lo que indica que ambas reacciones tienen lugar dentro del núcleo. Antes de que el miARN vegetal: los dúplex de miARN * se transporten fuera del núcleo, sus salientes 3 'son metilados por una proteína de ARN metiltransferasa llamada Hua-Enhancer1 (HEN1). Luego, el dúplex es transportado fuera del núcleo al citoplasma por una proteína llamada Hasty (HST), un homólogo de Exportin 5, donde se desmontan y el miARN maduro se incorpora al RISC.
Complejo silenciador inducido por ARN
El miARN maduro es parte de un complejo silenciador inducido por ARN activo (RISC) que contiene Dicer y muchas proteínas asociadas. RISC también se conoce como complejo de ribonucleoproteína de microARN (miRNP); Un RISC con miARN incorporado a veces se denomina "miRISC".
Se cree que el procesamiento en dicer del pre-miARN está acoplado con el desenrollado del dúplex. Generalmente, solo se incorpora una hebra en el miRISC, seleccionada sobre la base de su inestabilidad termodinámica y un apareamiento de bases más débil en el extremo 5 'en relación con la otra hebra. La posición del tallo-bucle también puede influir en la elección de la hebra. La otra hebra, llamada hebra pasajera debido a sus niveles más bajos en el estado estacionario, se indica con un asterisco (*) y normalmente está degradada. En algunos casos, ambas cadenas del dúplex son viables y se convierten en miARN funcionales que se dirigen a diferentes poblaciones de ARNm.
Los miembros de la familia de proteínas Argonaute (Ago) son fundamentales para la función RISC. Los argonautas son necesarios para el silenciamiento inducido por miARN y contienen dos dominios de unión de ARN conservados: un dominio PAZ que puede unirse al extremo 3 'monocatenario del miARN maduro y un dominio PIWI que estructuralmente se asemeja a la ribonucleasa-H y funciona para interactuar con el 5' extremo de la hebra guía. Se unen al miARN maduro y lo orientan para interactuar con un ARNm diana. Algunos argonautes, por ejemplo Ago2 humano, escinden las transcripciones objetivo directamente; los argonautes también pueden reclutar proteínas adicionales para lograr la represión traslacional. El genoma humano codifica ocho proteínas argonauta divididas por similitudes de secuencia en dos familias: AGO (con cuatro miembros presentes en todas las células de mamíferos y llamados E1F2C / hAgo en humanos) y PIWI (que se encuentra en la línea germinal y en las células madre hematopoyéticas).
Los componentes adicionales de RISC incluyen TRBP [proteína de unión al ARN de respuesta transactivante (TAR) del virus de inmunodeficiencia humana (VIH)], PACT (proteína activadora de la proteína quinasa inducida por interferón ), el complejo SMN, proteína de retraso mental X frágil (FMRP), estafilococo Tudor proteína que contiene el dominio de nucleasa (Tudor-SN), la supuesta ADN helicasa MOV10 y el motivo de reconocimiento de ARN que contiene la proteína TNRC6B .
Modo de silenciamiento y bucles reguladores
El silenciamiento de genes puede ocurrir a través de la degradación del ARNm o evitando que el ARNm se traduzca. Por ejemplo, miR16 contiene una secuencia complementaria al elemento rico en AU que se encuentra en el 3'UTR de muchos ARNm inestables, como TNF alfa o GM-CSF . Se ha demostrado que, dada la complementariedad completa entre el miARN y la secuencia de ARNm diana, Ago2 puede escindir el ARNm y conducir a la degradación directa del ARNm. En ausencia de complementariedad, el silenciamiento se logra evitando la traducción. La relación de miARN y su ARNm diana puede basarse en la simple regulación negativa de un ARNm diana, pero parece que un escenario común es el uso de un " bucle de retroalimentación coherente ", "bucle de retroalimentación negativa mutua" (también denominado bucle negativo doble) y "bucle de retroalimentación positiva / retroalimentación". Algunos miARN funcionan como amortiguadores de cambios aleatorios en la expresión génica que surgen debido a eventos estocásticos en la transcripción, traducción y estabilidad de las proteínas. Tal regulación se logra típicamente en virtud de bucles de retroalimentación negativa o salida de proteína de desacoplamiento de bucle de alimentación hacia adelante incoherente de la transcripción de ARNm.
Rotación
La rotación de miARN maduro es necesaria para cambios rápidos en los perfiles de expresión de miARN. Durante la maduración del miARN en el citoplasma, se cree que la captación de la proteína Argonaute estabiliza la hebra guía, mientras que la hebra opuesta (* o "pasajera") se destruye preferentemente. En lo que se ha llamado una estrategia de "Úselo o piérdalo", Argonaute puede retener preferentemente miARN con muchos objetivos sobre miARN con pocos o ningún objetivo, lo que lleva a la degradación de las moléculas no dirigidas.
Decay de maduro miRNAs en Caenorhabditis elegans está mediada por la 3' 5 'a exoribonuclease XRN2 , también conocido como Rat1p. En las plantas, los miembros de la familia SDN (nucleasa degradadora de ARN pequeño) degradan los miARN en la dirección opuesta (3 'a 5'). Enzimas similares están codificadas en genomas animales, pero sus funciones no se han descrito.
Varias modificaciones de miARN afectan la estabilidad de miARN. Como indica el trabajo en el organismo modelo Arabidopsis thaliana (thale berro), los miARN de plantas maduras parecen estabilizarse mediante la adición de restos metilo en el extremo 3 '. Los grupos metilo conjugados con 2'-O bloquean la adición de residuos de uracilo (U) por las enzimas uridiltransferasas , una modificación que puede estar asociada con la degradación de miARN. Sin embargo, la uridilación también puede proteger algunos miARN; las consecuencias de esta modificación se comprenden de forma incompleta. Se ha informado de la uridilación de algunos miARN de animales. Los miARN tanto de plantas como de animales pueden alterarse mediante la adición de residuos de adenina (A) al extremo 3 'del miARN. Un extra A añadido al final del miR-122 de mamíferos , un miARN enriquecido en hígado importante en la hepatitis C , estabiliza la molécula y los miARN de plantas que terminan con un residuo de adenina tienen tasas de descomposición más lentas.
Funciones celulares

La función de los miARN parece estar en la regulación genética. Para ese propósito, un miARN es complementario a una parte de uno o más ARN mensajeros (ARNm). Los miARN animales suelen ser complementarios a un sitio en la UTR 3 ', mientras que los miARN vegetales suelen ser complementarios a las regiones codificantes de los ARNm. El emparejamiento de bases perfecto o casi perfecto con el ARN objetivo promueve la escisión del ARN. Este es el modo principal de los miARN de plantas. En los animales, los enfrentamientos son imperfectos.
Para que los microARN parcialmente complementarios reconozcan sus objetivos, los nucleótidos 2 a 7 del miARN (su "región semilla") deben ser perfectamente complementarios. Los miARN animales inhiben la traducción de proteínas del ARNm diana (esto está presente pero es menos común en las plantas). Los microARN parcialmente complementarios también pueden acelerar la desadenilación , lo que hace que los ARNm se degraden antes. Si bien la degradación del ARNm dirigido a miARN está bien documentada, se debate acaloradamente si la represión de la traducción se logra a través de la degradación del ARNm, la inhibición de la traducción o una combinación de las dos. Un trabajo reciente sobre miR-430 en pez cebra, así como sobre bantam-miRNA y miR-9 en células cultivadas de Drosophila , muestra que la represión traduccional es causada por la interrupción del inicio de la traducción , independientemente de la desadenilación del mRNA.
Los miARN ocasionalmente también causan modificación de histonas y metilación del ADN de sitios promotores , lo que afecta la expresión de genes diana.
Se describen y ensamblan nueve mecanismos de acción de miARN en un modelo matemático unificado:
- Inhibición de la iniciación de Cap-40S;
- Inhibición de la unión de la unidad ribosómica 60S;
- Inhibición del alargamiento;
- Caída de ribosomas (terminación prematura);
- Degradación de proteínas nacientes co-traduccionales;
- Secuestro en cuerpos P;
- Decaimiento del ARNm (desestabilización);
- escisión de ARNm;
- Inhibición transcripcional a través de la reorganización de la cromatina mediada por microARN seguida de silenciamiento génico.
A menudo es imposible discernir estos mecanismos utilizando datos experimentales sobre velocidades de reacción estacionarias. Sin embargo, están diferenciados en dinámica y tienen diferentes firmas cinéticas .
A diferencia de los microARN de plantas, los microARN de animales se dirigen a diversos genes. Sin embargo, los genes implicados en funciones comunes a todas las células, como la expresión génica, tienen relativamente menos sitios diana de microARN y parecen estar bajo selección para evitar la orientación de microARN.
El dsRNA también puede activar la expresión génica , un mecanismo que se ha denominado "activación génica inducida por ARN pequeño" o ARNa . Los dsRNA que se dirigen a los promotores de genes pueden inducir una potente activación transcripcional de genes asociados. Esto se demostró en células humanas utilizando dsRNA sintéticos denominados RNA activadores pequeños ( saRNA ), pero también se ha demostrado para microRNA endógenos.
Se cree que las interacciones entre microARN y secuencias complementarias en genes e incluso pseudogenes que comparten homología de secuencia son un canal de comunicación que regula los niveles de expresión entre genes parálogos (genes que tienen una estructura similar que indica divergencia de un gen ancestral común). Dado el nombre de "ARN endógenos competidores" ( ARNc ), estos microARN se unen a "elementos de respuesta de microARN" en genes y pseudogenes y pueden proporcionar otra explicación para la persistencia del ADN no codificante.
Algunas investigaciones muestran que la carga de ARNm de los exosomas puede tener un papel en la implantación, pueden provocar una adhesión salvaje entre el trofoblasto y el endometrio o apoyar la adhesión regulando negativamente o regulando al alza la expresión de genes implicados en la adhesión / invasión.
Además, el miARN como miR-183/96/182 parece jugar un papel clave en el ritmo circadiano .
Evolución
Los miARN están bien conservados tanto en plantas como en animales, y se cree que son un componente vital y evolutivamente antiguo de la regulación genética. Si bien los componentes centrales de la vía del microARN se conservan entre plantas y animales , los repertorios de miARN en los dos reinos parecen haber surgido de forma independiente con diferentes modos de acción primarios.
Los microARN son marcadores filogenéticos útiles debido a su aparentemente baja tasa de evolución. El origen de los microARN como mecanismo regulador desarrollado a partir de la maquinaria de ARNi anterior que se utilizó inicialmente como defensa contra material genético exógeno como los virus. Su origen puede haber permitido el desarrollo de la innovación morfológica y, al hacer que la expresión génica sea más específica y "sintonizable", permitió la génesis de órganos complejos y quizás, en última instancia, la vida compleja. Las ráfagas rápidas de innovación morfológica generalmente se asocian con una alta tasa de acumulación de microARN.
Los nuevos microARN se crean de múltiples formas. Los nuevos microARN pueden originarse a partir de la formación aleatoria de horquillas en secciones "no codificantes" de ADN (es decir, intrones o regiones intergénicas), pero también mediante la duplicación y modificación de microARN existentes. Los microARN también pueden formarse a partir de duplicaciones invertidas de secuencias codificantes de proteínas, lo que permite la creación de una estructura de horquilla plegable. La tasa de evolución (es decir, sustitución de nucleótidos) en microARN de origen reciente es comparable a la de otras partes del ADN no codificante, lo que implica evolución por deriva neutra; sin embargo, los microARN más antiguos tienen una tasa de cambio mucho menor (a menudo menos de una sustitución por cada cien millones de años), lo que sugiere que una vez que un microARN adquiere una función, se somete a una selección purificadora. Las regiones individuales dentro de un gen de miARN enfrentan diferentes presiones evolutivas, donde las regiones que son vitales para el procesamiento y la función tienen niveles más altos de conservación. En este punto, un microARN rara vez se pierde del genoma de un animal, aunque los microARN más nuevos (por lo tanto, presumiblemente no funcionales) se pierden con frecuencia. En Arabidopsis thaliana , se ha previsto que el flujo neto de genes de miARN esté entre 1,2 y 3,3 genes por millón de años. Esto los convierte en un valioso marcador filogenético y se los considera una posible solución a problemas filogenéticos pendientes, como las relaciones de los artrópodos . Por otro lado, en múltiples casos, los microARN se correlacionan mal con la filogenia, y es posible que su concordancia filogenética refleje en gran medida una muestra limitada de microARN.
Los microARN se encuentran en los genomas de la mayoría de los organismos eucariotas, desde las algas pardas hasta los animales. Sin embargo, la diferencia en cómo funcionan estos microARN y la forma en que se procesan sugiere que los microARN surgieron de forma independiente en plantas y animales.
Centrándonos en los animales, el genoma de Mnemiopsis leidyi parece carecer de microARN reconocibles, así como de las proteínas nucleares Drosha y Pasha , que son fundamentales para la biogénesis de microARN canónicos. Es el único animal hasta ahora reportado que ha perdido a Drosha. Los microARN juegan un papel vital en la regulación de la expresión génica en todos los animales no ctenóforos investigados hasta ahora, excepto Trichoplax adhaerens , el único miembro conocido del filo Placozoa .
En todas las especies, en marzo de 2010 se habían identificado más de 5000 miARN diferentes. Mientras que en las bacterias se producen secuencias cortas de ARN (50 - cientos de pares de bases) de una función ampliamente comparable, las bacterias carecen de microARN verdaderos.
Detección y manipulación experimentales
Si bien los investigadores se centraron en la expresión de miARN en procesos fisiológicos y patológicos, surgieron varias variables técnicas relacionadas con el aislamiento de microARN. Se ha cuestionado la estabilidad de las muestras de miARN almacenadas. Los microARN se degradan mucho más fácilmente que los ARNm, en parte debido a su longitud, pero también debido a las ARNasas presentes de forma ubicua . Esto hace que sea necesario enfriar las muestras en hielo y utilizar equipos libres de ARNasa .
La expresión de microARN se puede cuantificar en un proceso de reacción en cadena de la polimerasa de dos pasos de RT-PCR modificada seguida de PCR cuantitativa . Las variaciones de este método logran una cuantificación absoluta o relativa. Los miARN también se pueden hibridar con microarrays , portaobjetos o chips con sondas para cientos o miles de objetivos de miARN, de modo que los niveles relativos de miARN se pueden determinar en diferentes muestras. Los microARN se pueden descubrir y perfilar mediante métodos de secuenciación de alto rendimiento ( secuenciación de microARN ). La actividad de un miARN puede inhibirse experimentalmente usando un oligo de ácido nucleico bloqueado (LNA) , un oligo Morfolino o un oligo 2'-O-metil ARN. Un miARN específico puede ser silenciado por un antagomir complementario . La maduración de microARN puede inhibirse en varios puntos mediante oligos de bloqueo estérico. El sitio diana de miARN de una transcripción de ARNm también puede bloquearse mediante un oligo de bloqueo estérico. Para la detección "in situ" de miARN, se pueden utilizar sondas de LNA o Morfolino. La conformación bloqueada de LNA da como resultado propiedades de hibridación mejoradas y aumenta la sensibilidad y la selectividad, lo que lo hace ideal para la detección de miARN cortos.
La cuantificación de alto rendimiento de miARN es propensa a errores, debido a la mayor variación (en comparación con los ARNm ) que conlleva problemas metodológicos. Por lo tanto, la expresión de ARNm se analiza a menudo para comprobar los efectos de miARN en sus niveles (por ejemplo, en). Las bases de datos se pueden utilizar para emparejar datos de ARNm y miARN que predicen objetivos de miARN en función de su secuencia de bases. Si bien esto generalmente se hace después de que se hayan detectado miARN de interés (por ejemplo, debido a altos niveles de expresión), se han propuesto ideas para herramientas de análisis que integran información de expresión de ARNm y miARN.
Enfermedad
Así como el miARN participa en el funcionamiento normal de las células eucariotas, la desregulación del miARN se ha asociado con la enfermedad. Una base de datos disponible públicamente y curada manualmente, miR2Disease, documenta las relaciones conocidas entre la desregulación de miARN y las enfermedades humanas.
Enfermedades hereditarias
Una mutación en la región de la semilla de miR-96 causa pérdida auditiva progresiva hereditaria.
Una mutación en la región de la semilla de miR-184 causa queratocono hereditario con catarata polar anterior.
La deleción del grupo miR-17 ~ 92 provoca defectos en el esqueleto y el crecimiento.
Cáncer
La primera enfermedad humana que se sabe que está asociada con la desregulación de miARN fue la leucemia linfocítica crónica. Muchos otros miARN también tienen vínculos con el cáncer y, en consecuencia, a veces se denominan " oncomirs ". En las células B malignas, los miARN participan en vías fundamentales para el desarrollo de las células B, como la señalización del receptor de células B (BCR), la migración / adhesión de células B, las interacciones célula-célula en nichos inmunes y la producción y cambio de clase de inmunoglobulinas. Los miARN influyen en la maduración de las células B, la generación de células B pre, zona marginal, folicular, B1, plasmática y de memoria.
Otro papel del miARN en los cánceres es utilizar su nivel de expresión para el pronóstico. En las muestras de NSCLC , los niveles bajos de miR-324 a pueden servir como indicador de una supervivencia deficiente. Los niveles altos de miR-185 o bajos de miR-133b pueden correlacionarse con metástasis y escasa supervivencia en el cáncer colorrectal .
Además, los miARN específicos pueden estar asociados con ciertos subtipos histológicos de cáncer colorrectal. Por ejemplo, se ha demostrado que los niveles de expresión de miR-205 y miR-373 aumentan en cánceres colorrectales mucinosos y cánceres de colon asociados a colitis ulcerosa productores de mucina, pero no en adenocarcinomas de colon esporádicos que carecen de componentes mucinosos. Los estudios in vitro sugirieron que miR-205 y miR-373 pueden inducir funcionalmente diferentes características de la progresión neoplásica asociada a mucinosas en las células epiteliales intestinales.
La proliferación de células de carcinoma hepatocelular puede surgir de la interacción de miR-21 con MAP2K3, un gen represor de tumores. El tratamiento óptimo para el cáncer implica identificar con precisión a los pacientes para la terapia estratificada por riesgo. Aquellos con una respuesta rápida al tratamiento inicial pueden beneficiarse de regímenes de tratamiento truncados, lo que demuestra el valor de las medidas precisas de respuesta a la enfermedad. Los miARN circulantes libres de células (cimiARN) son muy estables en sangre, se sobreexpresan en el cáncer y son cuantificables en el laboratorio de diagnóstico. En el linfoma de Hodgkin clásico , miR-21, miR-494 y miR-1973 en plasma son biomarcadores de respuesta a la enfermedad prometedores. Los miARN circulantes tienen el potencial de ayudar a la toma de decisiones clínicas y ayudar a la interpretación de la tomografía por emisión de positrones combinada con la tomografía computarizada . Se pueden realizar en cada consulta para evaluar la respuesta a la enfermedad y detectar recaídas.
Los microARN tienen el potencial de usarse como herramientas u objetivos para el tratamiento de diferentes cánceres. Se ha descubierto que el microARN específico, miR-506, actúa como antagonista tumoral en varios estudios. Se encontró que un número significativo de muestras de cáncer de cuello uterino tenían la expresión de miR-506 regulada negativamente. Además, miR-506 trabaja para promover la apoptosis de las células de cáncer de cuello uterino, a través de su factor de transcripción de la vía hedgehog objetivo directo, Gli3.
Reparación de ADN y cáncer
El cáncer es causado por la acumulación de mutaciones por daño del ADN o errores no corregidos en la replicación del ADN . Los defectos en la reparación del ADN provocan la acumulación de mutaciones que pueden provocar cáncer. Varios genes implicados en la reparación del ADN están regulados por microARN.
Las mutaciones de la línea germinal en los genes de reparación del ADN causan sólo del 2 al 5% de los casos de cáncer de colon . Sin embargo, la expresión alterada de microARN, que causa deficiencias en la reparación del ADN, se asocia frecuentemente con cánceres y puede ser un factor causal importante. Entre 68 cánceres de colon esporádicos con expresión reducida de la proteína MLH1 de reparación de desajustes de ADN , se encontró que la mayoría eran deficientes debido a la metilación epigenética de la isla CpG del gen MLH1 . Sin embargo, hasta el 15% de las deficiencias de MLH1 en cánceres de colon esporádicos parecen deberse a la sobreexpresión del microARN miR-155, que reprime la expresión de MLH1.
En 29 a 66% de los glioblastomas , la reparación del ADN es deficiente debido a la metilación epigenética del gen MGMT , que reduce la expresión proteica de MGMT. Sin embargo, para el 28% de los glioblastomas, la proteína MGMT es deficiente, pero el promotor MGMT no está metilado. En glioblastomas sin promotores de MGMT metilados, el nivel de microARN miR-181d se correlaciona inversamente con la expresión de proteínas de MGMT y el objetivo directo de miR-181d es el ARNm de MGMT 3'UTR (las tres regiones principales no traducidas del ARNm de MGMT). Por tanto, en el 28% de los glioblastomas, la expresión aumentada de miR-181d y la expresión reducida de la enzima de reparación del ADN MGMT pueden ser un factor causal.
Las proteínas HMGA (HMGA1a, HMGA1b y HMGA2) están implicadas en el cáncer y la expresión de estas proteínas está regulada por microARN. La expresión de HMGA es casi indetectable en tejidos adultos diferenciados, pero está elevada en muchos cánceres. Las proteínas HMGA son polipéptidos de ~ 100 residuos de aminoácidos caracterizados por una organización de secuencia modular. Estas proteínas tienen tres regiones altamente cargadas positivamente, denominadas ganchos AT , que se unen al surco menor de tramos de ADN ricos en AT en regiones específicas de ADN. Las neoplasias humanas, incluidos los carcinomas tiroideo, prostático, cervical, colorrectal, pancreático y ovárico, muestran un fuerte aumento de las proteínas HMGA1a y HMGA1b. Los ratones transgénicos con HMGA1 dirigido a células linfoides desarrollan linfoma agresivo, lo que demuestra que una alta expresión de HMGA1 está asociada con cánceres y que HMGA1 puede actuar como un oncogén. La proteína HMGA2 se dirige específicamente al promotor de ERCC1 , reduciendo así la expresión de este gen de reparación del ADN. La expresión de la proteína ERCC1 fue deficiente en el 100% de los 47 cánceres de colon evaluados (aunque se desconoce hasta qué punto estaba involucrado HGMA2).
Los polimorfismos de nucleótido único (SNP) pueden alterar la unión de miARN en 3'UTR, por ejemplo, el caso de hsa-mir181a y hsa-mir181b en el gen supresor de tumores CDON.
Enfermedad del corazón
El papel global de la función de miARN en el corazón se ha abordado inhibiendo condicionalmente la maduración de miARN en el corazón murino . Esto reveló que los miARN juegan un papel fundamental durante su desarrollo. Los estudios de perfiles de expresión de miARN demuestran que los niveles de expresión de miARN específicos cambian en corazones humanos enfermos, lo que apunta a su participación en miocardiopatías . Además, los estudios en animales sobre miARN específicos identificaron roles distintos para los miARN tanto durante el desarrollo del corazón como en condiciones patológicas, incluida la regulación de factores clave importantes para la cardiogénesis, la respuesta de crecimiento hipertrófico y la conductancia cardíaca. Otro papel de los miARN en las enfermedades cardiovasculares es utilizar sus niveles de expresión para el diagnóstico, pronóstico o estratificación del riesgo. Los miARN en modelos animales también se han relacionado con el metabolismo y la regulación del colesterol.
miARN-712
El microARN-712 murino es un biomarcador potencial (es decir, predictor) de la aterosclerosis , una enfermedad cardiovascular de la pared arterial asociada con la retención de lípidos y la inflamación. El flujo sanguíneo no laminar también se correlaciona con el desarrollo de aterosclerosis, ya que los mecanosores de las células endoteliales responden a la fuerza cortante del flujo alterado (flujo d). Varios genes proaterogénicos, incluidas las metaloproteinasas de matriz (MMP), están regulados positivamente por el flujo d, que median señales proinflamatorias y proangiogénicas. Estos hallazgos se observaron en arterias carótidas ligadas de ratones para imitar los efectos del d-flow. En 24 horas, miR-712 inmaduro preexistente formó miR-712 maduro, lo que sugiere que el miR-712 es sensible al flujo. Coincidiendo con estos resultados, miR-712 también se regula al alza en las células endoteliales expuestas al flujo d natural en la curvatura mayor del arco aórtico.
Origen
La secuencia de pre-ARNm de miR-712 se genera a partir del gen RN45s ribosómico murino en la región espaciadora interna transcrita 2 (ITS2). XRN1 es una exonucleasa que degrada la región ITS2 durante el procesamiento de RN45. Por tanto, la reducción de XRN1 en condiciones de d-flow conduce a la acumulación de miR-712.
Mecanismo
MiR-712 se dirige al inhibidor tisular de las metaloproteinasas 3 (TIMP3). Los TIMP normalmente regulan la actividad de las metaloproteinasas de la matriz (MMP) que degradan la matriz extracelular (MEC). La ECM arterial se compone principalmente de fibras de colágeno y elastina , que proporcionan el soporte estructural y las propiedades de retroceso de las arterias. Estas fibras juegan un papel fundamental en la regulación de la inflamación vascular y la permeabilidad, que son importantes en el desarrollo de la aterosclerosis. Expresado por células endoteliales, TIMP3 es el único TIMP unido a ECM. Una disminución en la expresión de TIMP3 da como resultado un aumento de la degradación de ECM en presencia de d-flow. De acuerdo con estos hallazgos, la inhibición de pre-miR712 aumenta la expresión de TIMP3 en las células, incluso cuando se exponen a un flujo turbulento.
TIMP3 también disminuye la expresión de TNFα (un regulador proinflamatorio) durante el flujo turbulento. La actividad de TNFα en flujo turbulento se midió mediante la expresión de la enzima convertidora de TNFα (TACE) en sangre. El TNFα disminuyó si se inhibía miR-712 o se sobreexpresaba TIMP3, lo que sugiere que miR-712 y TIMP3 regulan la actividad de TACE en condiciones de flujo turbulento.
Anti-miR-712 suprime eficazmente la expresión de miR-712 inducida por d-flow y aumenta la expresión de TIMP3. Anti-miR-712 también inhibe la hiperpermeabilidad vascular, lo que reduce significativamente el desarrollo de lesiones de aterosclerosis y la infiltración de células inmunes.
MicroARN-205 homólogo humano
El homólogo humano de miR-712 se encontró en el gen homólogo de RN45, que mantiene miARN similares a los de los ratones. MiR-205 de humanos comparte secuencias similares con miR-712 de ratones y se conserva en la mayoría de los vertebrados. MiR-205 y miR-712 también comparten más del 50% de los objetivos de señalización celular, incluido TIMP3.
Cuando se probó, d-flow disminuyó la expresión de XRN1 en humanos como lo hizo en las células endoteliales de ratones, lo que indica un papel potencialmente común de XRN1 en humanos.
Enfermedad del riñon
La deleción dirigida de Dicer en las células progenitoras renales derivadas de FoxD1 en un modelo murino dio como resultado un fenotipo renal complejo que incluía expansión de los progenitores de nefrona , menos células de renina , arteriolas del músculo liso , pérdida mesangial progresiva y aneurismas glomerulares. El perfil de transcriptoma completo de alto rendimiento del modelo de ratón knockout FoxD1-Dicer reveló una regulación positiva ectópica del gen proapoptótico, Bcl2L11 (Bim) y una desregulación de la vía p53 con aumento en los genes efectores de p53, incluidos Bax , Trp53inp1 , Jun, Cdkn1a , Mmp2 y Arid3a . Los niveles de proteína p53 permanecieron sin cambios, lo que sugiere que los miARN estromales de FoxD1 reprimen directamente los genes efectores de p53. Utilizando un enfoque de rastreo de linaje seguido de clasificación de células activadas por fluorescencia , el perfil de miARN de las células derivadas de FoxD1 no solo definió de manera integral el panorama transcripcional de los miARN que son críticos para el desarrollo vascular, sino que también identificó miARN clave que probablemente modularán el fenotipo renal. en su ausencia. Estos miARN incluyen miRs ‐ 10a, 18a, 19b, 24, 30c, 92a, 106a, 130a, 152, 181a, 214, 222, 302a, 370 y 381 que regulan Bcl2L11 (Bim) y miRs ‐ 15b, 18a, 21, 30c, 92a, 106a, 125b ‐ 5p, 145, 214, 222, 296‐5p y 302a que regulan los genes efectores de p53. De acuerdo con los resultados del perfil, se observó apoptosis ectópica en los derivados celulares del linaje progenitor derivado de FoxD1 y reitera la importancia de los miARN del estroma renal en la homeostasis celular.
Sistema nervioso
Los miARN parecen regular el desarrollo y la función del sistema nervioso . MiRNAs neuronales están involucrados en diferentes etapas de desarrollo sináptico, incluyendo dendritogénesis (que implica miR-132 , miR-134 y miR-124 ), sinapsis formación y maduración de la sinapsis (donde miR-134 y miR-138 se cree que está involucrado). La eliminación de la formación de miARN en ratones mediante el silenciamiento experimental de Dicer ha dado lugar a resultados patológicos, como reducción del tamaño neuronal y anomalías motoras cuando se silencia en las neuronas estriatales y neurodegeneración cuando se silencia en las neuronas del prosencéfalo . Algunos estudios encuentran expresión alterada de miARN en la enfermedad de Alzheimer , así como en esquizofrenia , trastorno bipolar , depresión mayor y trastornos de ansiedad .
Carrera
Según el Centro para el Control y la Prevención de Enfermedades, el accidente cerebrovascular es una de las principales causas de muerte y discapacidad a largo plazo en Estados Unidos. El 87% de los casos son accidentes cerebrovasculares isquémicos, que resultan del bloqueo en la arteria del cerebro que transporta sangre rica en oxígeno. La obstrucción del flujo sanguíneo significa que el cerebro no puede recibir los nutrientes necesarios, como oxígeno y glucosa, y eliminar desechos, como el dióxido de carbono. Los miARN juegan un papel en el silenciamiento de genes postraduccionales al dirigirse a genes en la patogénesis de la isquemia cerebral, como la vía inflamatoria, angiogénesis y apoptótica.
Alcoholismo
El papel vital de los miARN en la expresión génica es importante para la adicción , específicamente el alcoholismo . El abuso crónico de alcohol da como resultado cambios persistentes en la función cerebral mediados en parte por alteraciones en la expresión genética . La regulación global de miARN de muchos genes posteriores se considera significativa con respecto a la reorganización o conexiones sinápticas o adaptaciones neuronales a largo plazo que implican el cambio de comportamiento desde el consumo de alcohol hasta la abstinencia y / o dependencia. Se ha descubierto que hasta 35 miARN diferentes están alterados en el cerebro post-mortem alcohólico, todos los cuales se dirigen a genes que incluyen la regulación del ciclo celular , la apoptosis , la adhesión celular , el desarrollo del sistema nervioso y la señalización celular . Se encontraron niveles alterados de miARN en la corteza prefrontal medial de ratones dependientes del alcohol, lo que sugiere el papel del miARN en la orquestación de los desequilibrios traslacionales y la creación de proteínas expresadas diferencialmente dentro de un área del cerebro donde probablemente se originan el comportamiento cognitivo complejo y la toma de decisiones .
Los miARN se pueden regular hacia arriba o hacia abajo en respuesta al consumo crónico de alcohol. La expresión de miR-206 aumentó en la corteza prefrontal de ratas dependientes del alcohol, dirigiéndose al factor de transcripción del factor neurotrófico derivado del cerebro ( BDNF ) y, en última instancia, reduciendo su expresión. El BDNF juega un papel crítico en la formación y maduración de nuevas neuronas y sinapsis, lo que sugiere una posible implicación en el crecimiento de la sinapsis / plasticidad sináptica en los consumidores de alcohol. Se encontró que miR-155, importante en la regulación de las respuestas de neuroinflamación inducidas por el alcohol , estaba regulado al alza, lo que sugiere el papel de la microglía y las citocinas inflamatorias en la fisiopatología del alcohol. Se encontró una regulación a la baja de miR-382 en el núcleo accumbens , una estructura en el prosencéfalo basal importante en la regulación de los sentimientos de recompensa que potencia los hábitos de motivación. miR-382 es el objetivo del receptor de dopamina D1 (DRD1), y su sobreexpresión da como resultado la regulación positiva de DRD1 y delta fosB , un factor de transcripción que activa una serie de eventos de transcripción en el núcleo accumbens que finalmente resultan en comportamientos adictivos. Alternativamente, la sobreexpresión de miR-382 dio como resultado una bebida atenuada y la inhibición de la regulación al alza de DRD1 y delta fosB en modelos de rata de alcoholismo, lo que demuestra la posibilidad de usar productos farmacéuticos dirigidos a miARN en los tratamientos.
Obesidad
Los miARN juegan un papel crucial en la regulación de los progenitores de células madre que se diferencian en adipocitos . Se examinaron estudios para determinar qué papel desempeñan las células madre pluripotentes en la adipogénesis en la línea de células estromales inmortalizadas derivadas de la médula ósea humana hMSC-Tert20. Se ha encontrado una expresión disminuida de miR-155 , miR-221 y miR-222 durante la programación adipogénica de hMSC tanto inmortalizadas como primarias, lo que sugiere que actúan como reguladores negativos de la diferenciación. Por el contrario, la expresión ectópica de los miARN 155 , 221 y 222 inhibió significativamente la adipogénesis y reprimió la inducción de los reguladores maestros PPARγ y CCAAT / proteína de unión a potenciador alfa ( CEBPA ). Esto allana el camino para posibles tratamientos genéticos para la obesidad.
Otra clase de miARN que regula la resistencia a la insulina , la obesidad y la diabetes es la familia let-7 . Let-7 se acumula en los tejidos humanos durante el transcurso del envejecimiento . Cuando let-7 se sobreexpresó ectópicamente para imitar el envejecimiento acelerado, los ratones se volvieron resistentes a la insulina y, por lo tanto, más propensos a la obesidad y la diabetes inducidas por una dieta alta en grasas . En contraste, cuando let-7 fue inhibido por inyecciones de antagonistas específicos de let-7 , los ratones se vuelven más sensibles a la insulina y notablemente resistentes a la obesidad y la diabetes inducidas por una dieta alta en grasas. La inhibición de let-7 no solo podría prevenir la obesidad y la diabetes, sino que también podría revertir y curar la afección. Estos hallazgos experimentales sugieren que la inhibición de let-7 podría representar una nueva terapia para la obesidad y la diabetes tipo 2.
Hemostasia
Los miARN también juegan un papel crucial en la regulación de cascadas enzimáticas complejas, incluido el sistema hemostático de coagulación sanguínea. Los estudios a gran escala sobre el direccionamiento funcional de miARN han descubierto recientemente dianas terapéuticas fundamentales en el sistema hemostático.
ARN no codificantes
Cuando el proyecto del genoma humano cartografió su primer cromosoma en 1999, se predijo que el genoma contendría más de 100.000 genes codificadores de proteínas. Sin embargo, solo se identificaron alrededor de 20.000. Desde entonces, el advenimiento de los enfoques bioinformáticos combinados con estudios de ordenamiento genómico que examinan el transcriptoma, la secuenciación sistemática de bibliotecas de ADNc de longitud completa y la validación experimental (incluida la creación de oligonucleótidos antisentido derivados de miARN llamados antagomir ) han revelado que muchas transcripciones no codifican proteínas. ARN, que incluye varios ARNsno y miARN.
Virus
Los microARN virales juegan un papel importante en la regulación de la expresión génica de genes virales y / o del huésped para beneficiar al virus. Por lo tanto, los miARN juegan un papel clave en las interacciones huésped-virus y en la patogenia de las enfermedades virales. Se cree que la expresión de los activadores de la transcripción por el ADN del herpesvirus-6 humano está regulada por el miARN viral.
Predicción de destino
Los miARN pueden unirse a transcripciones de ARN mensajero (ARNm) diana de genes que codifican proteínas y controlar negativamente su traducción o causar la degradación del ARNm. Es de vital importancia identificar con precisión los objetivos de miARN. Está disponible una comparación del rendimiento predictivo de dieciocho algoritmos in silico . Los estudios a gran escala de la selección de miARN funcional sugieren que los algoritmos de predicción de la diana pueden pasar por alto muchos miARN funcionales.
Ver también
- Oligonucleótidos anti-miARN
- La expresion genica
- Lista de herramientas de predicción de genes de miARN
- Lista de herramientas de predicción de objetivos de miARN
- MicroADN
- miR-324-5p
- Interferencia de ARN
- Pequeño ARN interferente
- MicroARN derivado de ARN nucleolar pequeño
Referencias
Otras lecturas
- Definición y clasificación de miARN: Ambros V, Bartel B, Bartel DP, Burge CB, Carrington JC, Chen X, Dreyfuss G, Eddy SR, Griffiths-Jones S, Marshall M, Matzke M, Ruvkun G, Tuschl T (marzo de 2003). "Un sistema uniforme para la anotación de microARN" . ARN . 9 (3): 277–9. doi : 10.1261 / rna.2183803 . PMC 1370393 . PMID 12592000 .
- Revisión científica de ARN pequeño: Baulcombe D (septiembre de 2002). "Eventos de ADN. Un microcosmos de ARN". Ciencia . 297 (5589): 2002–3. doi : 10.1126 / science.1077906 . PMID 12242426 . S2CID 82531727 .
- Descubrimiento de lin-4 , el primer miARN descubierto: Lee RC, Feinbaum RL, Ambros V (diciembre de 1993). "El gen heterocrónico de C. elegans lin-4 codifica pequeños ARN con complementariedad antisentido con lin-14" . Celular . 75 (5): 843–54. doi : 10.1016 / 0092-8674 (93) 90529-Y . PMID 8252621 .
enlaces externos
- La base de datos miRBase
- miRTarBase , la base de datos de interacciones microARN-objetivo validada experimentalmente.
- semirna , aplicación web para la búsqueda de microARN en el genoma de una planta.
- ONCO.IO : Recurso integrador para el análisis de microARN y factores de transcripción en cáncer.
- MirOB : MicroRNA se dirige a la base de datos y la herramienta de análisis de datos y visualización de datos.
- Base de datos ChIPBase : una base de datos de acceso abierto para decodificar los factores de transcripción que estuvieron involucrados o afectaron la transcripción de microARN a partir de datos de ChIP-seq.
- Un video animado del proceso de biogénesis de microARN .
- Reactivos de modulación de miARN para permitir la regulación positiva o la supresión de la función de microARN maduro endógeno.