Vigilancia de ARNm - mRNA surveillance
Los mecanismos de vigilancia del ARNm son vías que utilizan los organismos para garantizar la fidelidad y la calidad de las moléculas de ARN mensajero (ARNm). Hay varios mecanismos de vigilancia presentes dentro de las células. Estos mecanismos funcionan en varios pasos de la ruta de biogénesis del ARNm para detectar y degradar las transcripciones que no se han procesado correctamente.
Visión general
La traducción de las transcripciones del ARN mensajero en proteínas es una parte vital del dogma central de la biología molecular . Sin embargo, las moléculas de ARNm son propensas a una serie de errores de fidelidad que pueden provocar errores en la traducción del ARNm en proteínas de calidad . Los mecanismos de vigilancia del ARN son métodos que utilizan las células para asegurar la calidad y fidelidad de las moléculas de ARNm. Esto se logra generalmente marcando una molécula de ARNm aberrante para su degradación por varias nucleasas endógenas .
Se ha documentado la vigilancia del ARNm en bacterias y levaduras . En eucariotas , se sabe que estos mecanismos funcionan tanto en el núcleo como en el citoplasma . Los controles de fidelidad de las moléculas de ARNm en el núcleo dan como resultado la degradación de las transcripciones procesadas incorrectamente antes de exportarlas al citoplasma. Las transcripciones están sujetas a más vigilancia una vez en el citoplasma. Los mecanismos de vigilancia citoplásmica evalúan las transcripciones de ARNm para detectar la ausencia o la presencia de codones de parada prematuros.
Actualmente se sabe que tres mecanismos de vigilancia funcionan dentro de las células : la vía de desintegración del ARNm mediada sin sentido (NMD); las vías de desintegración del ARNm mediadas ininterrumpidamente (NSD); y la vía de desintegración del ARNm mediada por no pasa (NGD).
Desintegración de ARNm mediada por tonterías
Visión general
La desintegración mediada por tonterías está involucrada en la detección y desintegración de las transcripciones de ARNm que contienen codones de terminación prematura (PTC). Los PTC pueden surgir en las células a través de varios mecanismos: mutaciones de la línea germinal en el ADN; mutaciones somáticas en el ADN; errores en la transcripción ; o errores en el procesamiento de ARNm postranscripcional. La falta de reconocimiento y descomposición de estas transcripciones de ARNm puede resultar en la producción de proteínas truncadas que pueden ser dañinas para el organismo. Al provocar la desintegración de los polipéptidos truncados en el extremo C , el mecanismo NMD puede proteger a las células contra los efectos deletéreos dominantes- negativos y de ganancia de función . Las PTC se han implicado en aproximadamente el 30% de todas las enfermedades hereditarias ; como tal, la vía NMD juega un papel vital para asegurar la supervivencia general y la aptitud de un organismo.
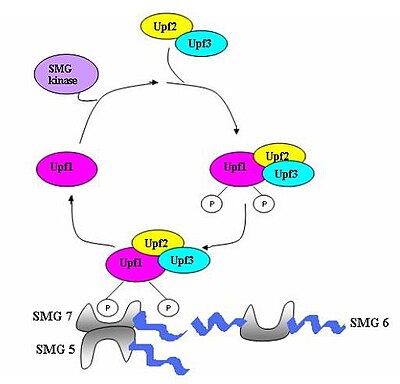
Se ensambla un complejo de vigilancia que consta de varias proteínas (eRF1, eRF3, Upf1, Upf2 y Upf3) y se escanea el ARNm en busca de codones de parada prematuros. El ensamblaje de este complejo se desencadena por la terminación prematura de la traducción. Si se detecta un codón de parada prematuro, se indica la degradación de la transcripción del ARNm: se produce el acoplamiento de la detección con la degradación.
Se han identificado siete genes smg (smg1-7) y tres genes UPF (Upf1-3) en Saccharomyces cerevisiae y Caenorhabditis elegans como factores esenciales de acción trans que contribuyen a la actividad de NMD. Todos estos genes se conservan en Drosophila melanogaster y otros mamíferos donde también desempeñan funciones críticas en la NMD. En todos los eucariotas hay tres componentes que se conservan en el proceso de NMD. Estos son los complejos Upf1 / SMG-2, Upf2 / SMG-3 y Upf3 / SMG-4. Upf1 / SMG-2 es una fosfoproteína en organismos multicelulares y se cree que contribuye a la NMD a través de su actividad de fosforilación. Sin embargo, actualmente se disputan las interacciones exactas de las proteínas y sus funciones en la NMD.
Mecanismo en mamíferos
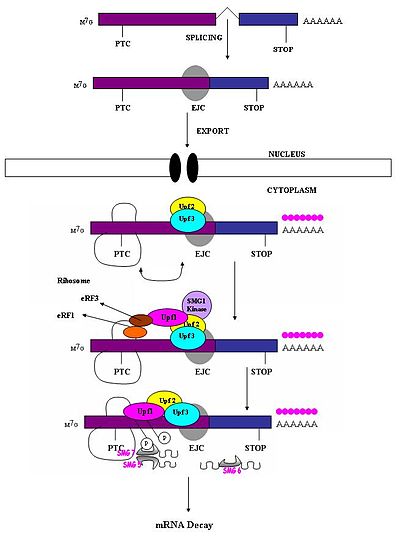
Un codón de terminación prematuro debe reconocerse como diferente de un codón de terminación normal de modo que solo el primero desencadene una respuesta NMD. Se ha observado que la capacidad de un codón sin sentido para provocar la degradación del ARNm depende de su ubicación relativa al elemento de secuencia aguas abajo y las proteínas asociadas. Los estudios han demostrado que los nucleótidos de más de 50 a 54 nucleótidos corriente arriba de la última unión exón-exón pueden dirigirse al ARNm para su desintegración. Los que se encuentran aguas abajo de esta región no pueden hacerlo. Por tanto, los codones sin sentido se encuentran a más de 50-54 nucleótidos cadena arriba del último límite del exón, mientras que los codones de parada naturales se encuentran dentro de los exones terminales. Los complejos de unión de exón (EJC) marcan los límites exón-exón. Los EJC son complejos multiproteicos que se ensamblan durante el empalme en una posición de aproximadamente 20 a 24 nucleótidos corriente arriba de la unión de empalme. Es este EJC el que proporciona la información de posición necesaria para discriminar los codones de parada prematuros de los codones de parada naturales. El reconocimiento de PTC parece depender de las definiciones de las uniones exón-exón. Esto sugiere la participación del espliceosoma en la NMD de mamíferos. La investigación ha investigado la posibilidad de participación del espliceosoma en la ENM de mamíferos y ha determinado que esta es una posibilidad probable. Además, se ha observado que los mecanismos de NMD no se activan mediante transcripciones sin sentido que se generan a partir de genes que naturalmente no contienen intrones (p. Ej., Histona H4, Hsp70, receptor de melanocortina-4).
Cuando el ribosoma alcanza un PTC, los factores de traducción eRF1 y eRF3 interactúan con los complejos EJC retenidos a través de un puente multiproteico. Las interacciones de UPF1 con el complejo de terminación y con UPF2 / UPF3 de los EJC retenidos son críticas. Son estas interacciones las que se dirigen al ARNm para una rápida descomposición por las nucleasas endógenas.

Mecanismo en invertebrados
Los estudios que involucran organismos como S. cerevisiae , D.melanogaster y C. elegans han demostrado que el reconocimiento de PTC que involucra organismos invertebrados no involucra límites exón-exón. Estos estudios sugieren que la NMD de invertebrados ocurre independientemente del empalme. Como resultado, los EJC que son responsables de marcar los límites exón-exón no son necesarios en la NMD de invertebrados. Se han propuesto varios modelos para explicar cómo se distinguen los PTC de los codones de parada normales en invertebrados. Uno de estos sugiere que puede haber un elemento de secuencia corriente abajo que funciona de manera similar a las uniones de exones en mamíferos. Un segundo modelo propone que una característica ampliamente presente en el ARNm, como una cola poli-A 3 ', podría proporcionar la información posicional requerida para el reconocimiento. Otro modelo, denominado "modelo falso 3'UTR", sugiere que la terminación prematura de la traducción puede distinguirse de la terminación normal debido a características intrínsecas que pueden permitirle reconocer su presencia en un entorno inapropiado. Sin embargo, estos mecanismos aún no se han demostrado de manera concluyente.
Mecanismo en plantas
Existen dos mecanismos de reconocimiento de PTC en plantas: según su distancia del EJC (como en los vertebrados) o de la cola poli-A. El mecanismo NMD en las plantas induce la descomposición de los ARNm que contienen un 3'UTR mayor de 300 nucleótidos, por eso la proporción de ARNm con 3'UTR más largos es mucho menor en las plantas que en los vertebrados.
Evitación de NMD
En general, se cree que los ARNm con mutaciones sin sentido son el objetivo de la desintegración a través de las vías de la NMD. La presencia de este codón de parada prematuro alrededor de 50-54 nucleótidos 5 'en la unión del exón parece ser el desencadenante de la desintegración rápida; sin embargo, se ha observado que algunas moléculas de ARNm con un codón de parada prematuro pueden evitar la detección y la descomposición. En general, estas moléculas de ARNm poseen el codón de terminación muy temprano en el marco de lectura (es decir, el PTC es proximal a AUG). Esto parece ser una contradicción con el modelo aceptado actual de NMD ya que esta posición es significativamente 5 'de la unión exón-exón.
Esto se ha demostrado en la β-globulina. Los ARNm de β-globulina que contienen una mutación sin sentido temprano en el primer exón del gen son más estables que las moléculas de ARNm sensibles a NMD. Actualmente se desconoce el mecanismo exacto para evitar la detección. Se ha sugerido que la proteína de unión poli-A (PABP) parece desempeñar un papel en esta estabilidad. Se ha demostrado en otros estudios que la presencia de esta proteína cerca de los PTC proximales a AUG parece promover la estabilidad de estos ARNm que de otro modo serían sensibles a NMD. Se ha observado que este efecto protector no se limita únicamente al promotor de la β-globulina. Esto sugiere que este mecanismo de evitación de NMD puede prevalecer en otros tipos de tejidos para una variedad de genes. Es posible que sea necesario revisar el modelo actual de NMD en estudios posteriores.
Desintegración de ARNm mediada sin parar
Visión general
La desintegración mediada ininterrumpida (NSD) está involucrada en la detección y desintegración de las transcripciones de ARNm que carecen de un codón de terminación. Estas transcripciones de ARNm pueden surgir de muchos mecanismos diferentes, como señales de adenilación 3 'prematura o poliadenilación críptica dentro de la región codificante de un gen. Esta falta de un codón de terminación resulta en un problema importante para las células. Los ribosomas que traducen el ARNm finalmente se traducen en la región de la cola 3'poly-A de transcripciones y paradas. Como resultado, no puede expulsar el ARNm. Por tanto, los ribosomas pueden quedar secuestrados asociados con el ARNm ininterrumpido y no estarían disponibles para traducir otras moléculas de ARNm en proteínas. La descomposición mediada ininterrumpidamente resuelve este problema liberando los ribosomas estancados y marcando el ARNm ininterrumpido para su degradación en la célula por las nucleasas. La desintegración mediada ininterrumpidamente consta de dos vías distintas que probablemente actúan en concierto para la desintegración ininterrumpida del ARNm.
Camino Ski7
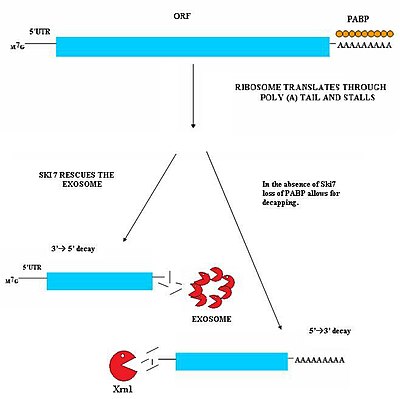
Esta vía está activa cuando la proteína Ski7 está disponible en la célula. Se cree que la proteína Ski7 se une al sitio A vacío del ribosoma. Esta unión permite que el ribosoma expulse la molécula de ARNm atascada sin parar; esto incluso libera el ribosoma y le permite traducir otras transcripciones. El Ski7 ahora está asociado con el ARNm ininterrumpido y es esta asociación la que se dirige al ARNm ininterrumpido para que lo reconozca el exosoma citosólico . El complejo Ski7-exosoma desadenila rápidamente la molécula de ARNm, lo que permite que el exosoma descomponga la transcripción en una forma de 3 'a 5'.
Camino sin esquí7
Se ha observado un segundo tipo de NSD en la levadura. En este mecanismo, la ausencia de Ski7 da como resultado la pérdida de proteínas PABP que se unen a la cola poli-A por la acción del ribosoma de traducción. La eliminación de estas proteínas PABP da como resultado la pérdida de la capa protectora de 5'm7G . La pérdida de la tapa da como resultado una rápida degradación del transcrito por una exonucleasa endógena 5'-3 'como XrnI.
Decaimiento no-Go
La descomposición No-Go (NGD) es el mecanismo de vigilancia descubierto más recientemente. Como tal, actualmente no se comprende bien. Si bien los objetivos auténticos de NGD no se conocen bien, parecen consistir principalmente en transcripciones de ARNm en las que los ribosomas se han estancado durante la traducción. Este estancamiento puede ser causado por una variedad de factores, incluidas estructuras secundarias fuertes , que pueden bloquear físicamente la maquinaria de traducción para que no se mueva hacia abajo en la transcripción. Dom34 / Hbs1 probablemente se une cerca del sitio A de los ribosomas estancados y puede facilitar el reciclaje de complejos. En algunos casos, la transcripción también se escinde de forma endonucleolítica cerca del sitio de bloqueo; sin embargo, la identidad de la endonucleasa responsable sigue siendo controvertida. Las moléculas de ARNm fragmentadas son luego completamente degradadas por el exosoma de una forma de 3 'a 5' y por Xrn1 de una forma de 5 'a 3'. Actualmente no se sabe cómo este proceso libera el ARNm de los ribosomas, sin embargo, Hbs1 está estrechamente relacionado con la proteína Ski7 que juega un papel claro en la liberación de ribosomas en la NSD mediada por Ski7. Se postula que Hbs1 puede desempeñar un papel similar en NGD.
Evolución
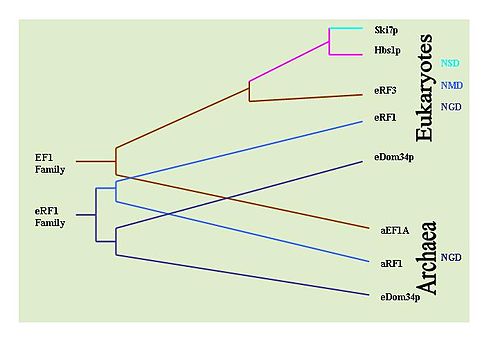
Es posible determinar la historia evolutiva de estos mecanismos observando la conservación de proteínas clave implicadas en cada mecanismo. Por ejemplo: Dom34 / Hbs1 están asociados con NGD; Ski7 está asociado con NSD; y las proteínas eRF están asociadas con NMD. Con este fin, se han realizado extensas búsquedas BLAST para determinar la prevalencia de las proteínas en varios tipos de organismos. Se ha determinado que NGD Hbs1 y NMD eRF3 se encuentran solo en eucariotas. Sin embargo, el NGD Dom34 es universal en eucariotas y arqueas . Esto sugiere que NGD parece haber sido el primer mecanismo de vigilancia de ARNm desarrollado. La proteína NSD Ski7 parece estar restringida estrictamente a las especies de levadura, lo que sugiere que la NSD es el mecanismo de vigilancia de evolución más reciente. Esto, por defecto, deja a NMD como el segundo mecanismo de vigilancia evolucionado.