Proteína que interactúa con el receptor de glutamato - Glutamate receptor-interacting protein
La proteína que interactúa con el receptor de glutamato (GRIP) se refiere a una familia de proteínas que se unen al receptor de glutamato o específicamente a la proteína GRIP1 dentro de esta familia. Se ha demostrado que las proteínas de la familia de proteínas que interactúan con el receptor de glutamato (GRIP) interactúan con GluR2 , una subunidad común en el receptor AMPA . Esta subunidad también interactúa con otras proteínas, como la proteína que interactúa con la C-quinasa1 ( PICK1 ) y la proteína de fusión sensible a N-etilmaleimida ( NSF ). Los estudios han comenzado a dilucidar su función; sin embargo, aún queda mucho por aprender sobre estas proteínas.
Descubrimiento e historia de GRIP 1
El descubrimiento de la proteína que interactúa con el receptor de glutamato (GRIP-1) se produjo como resultado de la observación de que los receptores de glutamato, como el receptor de NMDA , se agrupan en las sinapsis. Poco después de esta observación, los investigadores identificaron una región en la región C-terminal de los receptores NMDA llamada motivo tSXV que tiene la capacidad de unirse al dominio PDZ de la proteína PSD-95 .
La investigación sobre la localización del receptor de NMDA allanó el camino para la investigación de receptores que no son de NMDA, como los receptores de AMPA. De forma similar a los receptores NMDA, se descubrió que los receptores AMPA se localizan en la terminal sináptica de las neuronas del sistema nervioso central . Mediante el uso de anticuerpos GFP ( proteína verde fluorescente ) que corresponden a la proteína GRIP, los investigadores pudieron usar la fluorescencia para determinar la ubicación de GRIP en las neuronas del hipocampo. Luego se usó otro anticuerpo GFP para marcar la subunidad GluR2 de los receptores AMPA. Mediante el uso de inmunocitoquímica y la comparación de la ubicación de los receptores GRIP y AMPA se determinó que los receptores GRIP y AMPA experimentan colocalización en las neuronas del hipocampo. Estos hallazgos confirmaron la hipótesis inicial de que la proteína GRIP juega un papel importante en la unión de los receptores AMPA a las sinapsis excitadoras.
La estructura de GRIP contiene siete dominios PDZ y se une al terminal C de la subunidad GluR2 de los receptores AMPA. Aunque el número de dominios PDZ es diferente para las proteínas PSD-95 y GRIP, el dominio PDZ es un motivo estructural común en proteínas que ayudan a mediar las interacciones proteína-proteína. La secuencia de aminoácidos del receptor AMPA a la que se une la proteína GRIP es ESVKI. El aminoácido serina conservado en el extremo C-terminal de los receptores AMPA y NMDA sugiere que juega un papel importante en la facilitación de la interacción para GRIP y PSD-95.
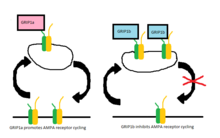
Papel de GRIP en el ciclismo AMPAR
Los receptores AMPA se transportan constantemente entre la membrana celular y el espacio intracelular y originalmente se pensó que GRIP podría ser responsable de la agrupación de receptores AMPA en la sinapsis excitadora. Aunque todavía no está claro el papel exacto de GRIP en este tráfico, parece que PICK1 es más directamente responsable de la agrupación de receptores AMPA en la superficie y que GRIP está involucrado en la estabilización de los receptores AMPA intracelularmente. Un estudio mostró que cuando se interrumpe la interacción entre GluR2 y GRIP, no hay cambios en la expresión superficial de los receptores AMPA o la internalización constitutiva de los receptores AMPA. Sin embargo, hay una cantidad reducida de receptores que permanecen internalizados cuando el ciclo del receptor se modifica mediante la aplicación de AMPA-1. La proporción vuelve a la normalidad cuando se permite que ocurra el reciclaje constitutivo, lo que sugiere que la estabilización de los receptores intracelulares es crítica solo bajo la internalización inducida por AMPA.
En estudios posteriores , se descubrió que dos proteínas, GRIP-1 (a menudo reducida a GRIP) y ABP-L (también llamada GRIP-2 ), se expresaban mediante dos genes separados y sus respectivas contribuciones al ciclo del receptor de AMPA se han estudiado bien desde entonces. . Cada una de estas proteínas tiene diferentes isoformas debido al empalme diferencial del ARN . Las isoformas de GRIP-1 se denominan GRIP-1a y GRIP-1b, mientras que las de ABP-L se distinguen como ABP-L y pABP-L. La diferencia aparente en ambos casos es que una isoforma (GRIP1b y pABP-L respectivamente) es capaz de conjugarse con ácido palmítico , una acción llamada palmitoilación .
Mientras que inicialmente se pensaba que GRIP estaba involucrado en la estabilización de los receptores de AMPA en la superficie celular o intracelularmente cuando la internalización se desencadenaba por la estimulación de AMPA, ahora parece que las isoformas de GRIP-1 están involucradas de manera diferencial con la estabilización de los receptores de AMPA después de ser internalizadas. debido a la estimulación NMDA . Se ha demostrado que GRIP-1a reduce los niveles intracelulares esperados de receptores AMPA después de la estimulación con NMDA. Por el contrario, GRIP-1b aumenta los niveles intracelulares de receptores AMPA en las mismas condiciones.
ABP-L, como GRIP-1b, se asocia con depósitos intracelulares de receptores AMPA. pABP-L, sin embargo, se asocia con los receptores AMPA como la membrana de superficie. Aún no se ha demostrado en qué condiciones estas interacciones son significativas en el ciclo de AMPAR.
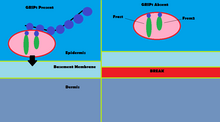
Papel de GRIP1 en el síndrome de Fraser
Las mutaciones en GRIP1 juegan un papel en menos del 10% de los casos confirmados del grupo de defectos congénitos conocido como síndrome de Fraser . Usando inmunofluorescencia , se ha demostrado que GRIP1 se encuentra en varios tipos de tejidos embrionarios, incluido el tracto gastrointestinal, yemas de uréter, piel y cavidades oral y nasal. GRIP1 también es esencial para el funcionamiento y la estructura adecuados de la unión dermoepidérmica. En modelos de ratón, la eliminación de la proteína GRIP1 conduce a varias deformidades que comienzan en el embrión. Estas deformidades incluyen ampollas hemorrágicas subepidérmicas, agenesia renal , sindactilismo , polidactilismo y criptopthalmos. Un estudio ha demostrado que la eliminación completa de GRIP1 conduce a la ausencia de riñones. Otro estudio muestra la formación de ampollas en el tejido embrionario en el que se expresa GRIP1 el día 12 de vida embrionaria en ratones.
El mecanismo de GRIP1 en el síndrome de Fraser se encuentra en la interacción que GRIP1 tiene con las proteínas Fras1 y Frem2. Fras1 y Frem2 son proteínas de la membrana extracelular necesarias para la función adecuada de la membrana basal y la morfogénesis . GRIP1 juega un papel vital en la localización de Fras1 en la superficie basal de las células epidérmicas, así como en la localización de Frem2. La eliminación de la proteína GRIP1 o su mutación conduce a una expresión deficiente de Fras1 y Frem2. GRIP1 se une específicamente con Fras1 a través de un motivo PDZ ubicado en Fras1. Frem2 también tiene un dominio PDZ , aunque la interacción entre GRIP1 y Frem2 no está clara. En un caso de síndrome de Fraser, GRIP1 carecía de los dominios 6 y 7 de PDZ. Solo los primeros cuatro dominios de PDZ de los siete dominios de PDZ que tiene GRIP1 son necesarios para unirse con Fras1, lo que indica mecanismos adicionales y proteínas con las que GRIP1 interactúa y que podrían conducir al síndrome de Fraser cuando mutado. Otras mutaciones en GRIP1 que conducen al síndrome de Fraser incluyen mutaciones sin sentido, mutaciones de cambio de marco, mutaciones en el sitio de empalme, una deleción del genoma y una deleción del exón 18 del gen GRIP1.
Papel de GRIP1 en la morfología neuronal y el transporte de carga
La morfología , el desarrollo y el mantenimiento de las neuronas dependen de la expresión de GRIP1 en la célula. Es de vital importancia en el desarrollo inicial, ya que los experimentos de knock-out en modelos murinos dan como resultado ampollas en la piel y letalidad embrionaria. En modelos murinos desarrollados, mutaciones incapacitantes como la transfección o los negativos dominantes en GRIP1 pueden causar hasta un 75% de pérdida de dendritas "primarias, secundarias y de orden superior" en las neuronas en desarrollo. La desactivación de GRIP1 en neuronas vivas sanas en un plato provocará una reducción del 20% en la parte más gruesa de la neurona y hasta un 70% de reducción en las ramas.
Se pueden reservar los defectos en la morfología neuronal debidos al mal funcionamiento de GRIP1. Una forma es sobreexpresar GRIP1. Esto conduce a una recuperación aumentada, pero no completa, de la ramificación. Otra proteína, EphB2 , que interactúa con GRIP1, se puede mutar de manera que sea posible una recuperación de la ramificación del 70-90%. Sin embargo, la sobreexpresión del tipo salvaje conduce a una disminución en el recuento de neuronas.
Las proteínas motoras como la kinesina (KIF5) están unidas a moléculas adaptadoras como GRIP1 para mover la carga desde el Golgi a las extremidades de una célula neuronal. GRIP1 y KIF5 se encuentran muy comúnmente juntos debido a una buena afinidad de unión (rango de Kd de 10-20 nM). En cuanto a cómo la carga llega al lugar correcto, existe una hipótesis llamada “motor inteligente”. Actualmente se piensa que el "motor inteligente" reconoce la diferencia entre las proteínas axonales (recubiertas con proteína KLC) y dendríticas (recubiertas con KHC). El destino se elige en consecuencia. Desafortunadamente, se desconocen los detalles sobre los pasos de transporte intermedios. Sin embargo, en el destino, la unión de la proteína 14-3-3 interrumpe la interacción entre KIF5 y GRIP1. Esto libera la carga.